
LABORATORIO DE BIOTECNOLOGÍA CELULAR Y MOLECULAR AVANZADA (LAB-BIOTCEMA)
- DESCRIPCIÓN: Laboratorio formado en la Universidad Nacional de San Agustín, Arequipa, Perú con el fin de explorar las ciencias básicas y aplicadas en diversos modelos biológicos.
- MISIÓN: Poder realizar estudios sobre biotecnología molecular en diferentes modelos biológicos, desde lo básico a la aplicación.
-INTEGRANTES
:-Dr. Antonio Lazarte Rivera (Director del proyecto de implementación, UNSA-Arequipa, Perú) PROYECTO COVID-19
-MSc. Ronald Huarachi Olivera (Asistente Externo de Investigación, Estudiante de Doctorado en Ciencias Biológicas con mención en Biología Celular y Molecular, Universidad de Antofagasta, Chile). PROYECTO COVID-19
- Danilo Joel Centeno Gonzales (Tesista de pregrado de la carrera de Biología de la Universidad Nacional de San Agustín, Arequipa, Perú).
-Kenyi Angelo, Arango Farias (Tesista pregrado de la carrera de Biología, Universidad Nacional de San Agustín, Arequipa, Perú. PROYECTO COVID-19
-Luis Ancca (tesista de la carrera de Biología, Universidad Nacional de San Agustín, Arequipa, Perú).
-Daniela Victoria Arequipa Loayza (tesista de la carrera de Biología, Universidad Nacional de San Agustín, Arequipa, Perú).
-David Huayhua Yauli (tesista de la carrera de Biología, Universidad Nacional de San Agustín, Arequipa, Perú).
-Orquidea Lucero
Merma Torres (tesista de la carrera de Biología, Universidad Nacional de San Agustín, Arequipa, Perú).
-Maria Quenta Chuctaya, (tesista de la carrera de Biología, Universidad Nacional de San Agustín, Arequipa, Perú).
-Carmen Rosario Yauri Chacòn (estudiante de la carrera de Biologia, Universidad Nacional de San Agustìn , Arequipa, Perù
-Liseth Orosco Barrionuevo (Tesista pregrado de la carrera de Biología, Universidad Nacional de San Agustín, Arequipa, Perú y Universidad San Sebastian , Chile). PROYECTO COVID-19
-Angel Francisco Solis Gozar (tesista Molecular de la Universidad Nacional Mayor de San Marcos, Lima Perù) PROYECTO COVID-19
.-Carlos Enrique Salcedo Ballon(estudiante de la carrera de Ciencias de Nutriciòn de la Universidad Nacional de San Agustín, Arequipa, Perù).PROYECTO COVID-19
-Jhon Brando Ortiz Saavedra (Estudiante de la carrera de Medicina de la Universidad Nacional de San Agustìn, Arequipa, Perù) PROYECTO COVID-19
Líneas de Investigación:
-Análisis múltiple de genes (Genómica Funcional).
-Sistemática Molecular.
-Ecología y Bioestadística Molecular.
-Fisiología y Biotecnología Molecular Vegetal.
-Bioinformática y Expresión Génica.
-Biología circadiana y ciclo celular.
-COVID-19.
Conferencias Magistrales y/o cursos realizados (LAB-BIOTCEMA)
SOFTWARE QUE UTILIZAMOS
1.- ImageLab 6.0 (Chemidoc)
https://drive.google.com/file/d/1tGQiSSM7zJvTeEAVXBL4VCCxUzeXyvPy/view?usp=sharing
2.- Quantasoft (QX200)
https://drive.google.com/file/d/1C7NPDSch6mq9RRfh0ZkbsW6zmN6EXG2v/view?usp=sharing
3.- MEGA
4.-Cytoscape 3.7.1
https://cytoscape.org/download.html
5.-Systems Biology
https://virtualplant.bio.puc.cl/cgi-bin/Lab/index.cgi
6.- Geneontology
https://amigo.geneontology.org/amigo
https://www.ebi.ac.uk/QuickGO/
7.- TSGene database
https://bioinfo.uth.edu//TSGene1.0/
8.- GraphPad online
https://www.graphpad.com/quickcalcs/contingency1/
9.-Conjunto
de datos de ARN completo para microarrays
10.-Datos ARN "GEO"
https://www.ncbi.nlm.nih.gov/geo/
11.-Archivos GPR depositados en el microarray del NCI, base de datos mAdb
12.- Gene Expression Universal Reference RNAs
13.-Pomelo2 para analizar LIMMA(linear analysis of microarrays data; análisis lineal de datos de microarreglos)
14.-WebaCGH: Permite la detección de ganancias y pérdidas cromosómicas en un formato de alta resolución
https://omictools.com/webacgh-tool
15.- Bajo el supuesto de que los niveles de transcripción son directamente proporcionales a los niveles de proteína, utilizamos el STRING v10.
https://string-db.org/cgi/input.pl
16.-DEG deben someterse a GO, KEGG y WikiPathway analisis en:
-webgestalt
-webgestalt (versión antigua)
https://www.webgestalt.org/2013/
-VLAD
https://proto.informatics.jax.org/prototypes/vlad/
17.-TCGA data
https://www.cancer.gov/about-nci/organization/ccg/research/structural-genomics/tcga/?redirect=true
18.- GenePix Pro 6.0
https://axon-genepix-pro.software.informer.com/6.0/
19.- HGNC (HUGO Gene Nomenclature Commitee)
20.- Genecard (donde estan descritos los ALIAS)
21.- 7184 NCBI (identificador múltiple de genes)
https://www.ncbi.nlm.nih.gov/gene/7184
22.- ENSG00000166598 (Alineamiento del genoma)
https://www.ensembl.org/index.html
23.- 191175
https://www.ncbi.nlm.nih.gov/omim
24.- 01860
25.- STRING DATABASE (interacciones proteína-proteína)
26.-GEO (Gene Expression Omnibus)
https://www.ncbi.nlm.nih.gov/geo/
27.-Para la anotación funcional de secuencias de Gene Ontology (GO) se usa el software Blast2go.
https://www.blast2go.com/blast2go-pro/download-b2g
28.-Los números de acceso de Gene Ontology(GO) se envia la herramienta de análisis WEGO (en línea) para obtener la clasificación funcional de GO
https://biodb.swu.edu.cn/cgi-bin/wego/index.pl
29.-Entrez Map Viewer Help Document (sitio que nos permite ver todos los genomas de diferentes lineas celulares descritos hasta ahora)
https://www.ncbi.nlm.nih.gov/projects/mapview/static/MapViewerHelp.html
https://www.ncbi.nlm.nih.gov/genome/gdv/
30.- Las lecturas de transcriptos seleccionados son normalizados usando Trimmed Mean of M-value(TMM), método de normalización, siendo analizados usando Multiple Experiment Viewer (MeV 4.8.1 version) software
31.-Las lecturas obtenidas por RNA-seq se deben recortar utilizando Prinseq (version 0.20.4; −min_len 50 -min_qual_mean 20-ns_max_n 1 -derep 14 -derep_min 9 -lc_method dust-lc_threshold 49 -trim_left 10 -trim_qual_right)
https://sourceforge.net/projects/prinseq/
32.- tBLASTn: Genera una secuencia nucleotídica a partir de una secuencia aminoacídica
https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=tblastn&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome
33.- KEGG ORTHOLOGY (KO).
https://www.genome.jp/dbget-bin/www_bget?ko:K06641
34.-El programa que nos permite ensamblar el Transcriptoma es el Trinity (/−-min_contig_length 200)
35.- El programa que nos permite mapear el transcriptoma de novo, ósea contar el número de reads es: Bowtie2 (version 2.2.9; default settings).
36.- Plataforma libre y gratuita del Linux
37.-Nuevas bibliotecas para genes, nos sirve para buscar los genes colocando sus simbolos
https://amp.pharm.mssm.edu/Enrichr/
38.- Empresa de secuenciamiento con el cual trabajamos
39.- SOFTWARE ANALIZADOS CON EL DR. ULISES URZUA, Universidad de Chile
39.1.- HGNC (HUGO Gene Nomenclature Commitee) https://www.genenames.org/
¿En
que contexto o problemática se aplican?
HGNC es la única autoridad científica a nivel mundial que asigna nomenclatura estandarizada a los genes humanos. Debido a que cada gen tiene su símbolo y nombre descriptivo, esta base de datos tiene un identificador HGNC ID.
Aplicación:
El HGNC ID nos sirve para distinguir un gen de otro gen cuando se trata de ortólogos, siendo los mismos genes que cumplen la misma función en distintas especies. Es decir, sirve para identificar una especie de otra especie. Sin embargo, este identificador no es muy usado, siendo más usado el identificador de NCBI, específicamente mediante el Entrez, que es un acceso integrado a datos de secuencia de nucleótidos y proteínas, información de mapeo genómico y centrada en el gen y los datos de estructura 3D, el cual proporciona distintos valores para distintas especies. .
39.2.- Genecard (donde están descritos los ALIAS) https://www.genecards.org/
¿En qué contexto o problemática
se aplican?
Software que nos permite encontrar directamente los alias de los genes de estudio, también nos brinda la información de los identificadores así como el HGNC y el ENTREZ, así como el Ensembl para observar la secuencia del gen de estudio.
39.3.- Gene Ontology (GO)
https://www.ebi.ac.uk/QuickGO/
¿En qué
contexto o problemática se aplican?
Es una Colección sistemática y jerárquica de conceptos y/o clases que mediante el uso de un vocabulario controlado describen a cada gen en términos de su: Proceso biológico, Componente celular y Función molecular basado en evidencia designado por códigos de:
1° Genes de estudios experimentales. 2° Genes de estudio del Proyecto Genoma. 3° Información trazable o no trazable través de un paper o texto)
Donde los códigos de los genes de estudios experimentales son los más fuertes y es lo que se desearía como resultado y en cuanto al 2° Hay genes que todavía no se sabe que hacen.
39.4. Genes expresados diferencialmente (DEG) deben someterse al análisis Gene Ontology(GO), Genes y genomas de la enciclopedia de Kyoto(KEGG) en:
Webgestalt
¿En qué contexto o problemática
se aplican?
Sirve para procesar una lista de un gran número genes almacenados en Excel en archivo TXT, de una especie en estudio, clasificando esa lista de genes en base a: Procesos Biológicos, función molecular y componente celular, a través del análisis de enriquecimiento de GeneOntology(GO), a la vez también nos brinda una información de los identificadores de cada gen mediante ENTREZ y el ensamble de cada gen con el ENSEMBL, indicando dentro de esa lista que genes son no codificantes.
Para el análisis del GO, trabajamos con una lista de genes en archivo en Excel (TXT), en Enrichment Analysis se presiona el GO y en GO Slim Classification se elije: procesos biológicos, función molecular y componente celular, y en base a esa elección nos da como resultado las categorías de: proceso biológico, función molecular y componente celular; donde la altura de la barra representa el número de genes de la lista de genes expresados diferencialmente observados en cada categoría.
FUENTE: https://www.webgestalt.org/2013/analysis.php
El GO tiene mayor cobertura a diferencia de la base de datos "Genes y genomas de la enciclopedia de Kyoto (KEGG)" siendo una web japonesa , es el más antiguo, el cual aún se sigue usando, el cual nos da la información de las vías metabólicas y bioquímicas que muestra las interacciones de un gen con otro gen. El KEGG analiza los siguientes procesos: Metabolismo, procesamiento de la información genética, procesamiento de la información ambiental, procesos celulares, sistemas de órganos y enfermedades humanas siendo una de las vías más grandes que existe, así como los target, enfermedades neurodegenerativas, enfermedades cardiovasculares, etc.. .
Para al análisis KEGG, en webgestalt, usamos el análisis de enriquecimiento, el cual elegimos una lista de genes de interés en Excel(archivo TXT), ya que nos determina las vías metabólicas bioquímicas, al realizar esta operación el programa webgestalt en base a unos estadístico como clasifica la lista de genes en tres categorías:
- C: el número de genes de referencia - O: el número de genes observados en el conjunto de genes. - E: el número de genes esperados
Es decir si se investiga en un modelo biológico de 20000 genes del genoma y en total hay 2000 genes asignados para esa función por ejemplo: proteing binding, ese valor sería el valor de (C) que tiene ese descrito de función molecular, y si la lista de genes expresados diferencialmente dicta 100 genes y 10 de ellos tienen función (binding protein) siendo el valor de (O), entonces la proporción global seria de 2000/20000 dando lugar al valor de (E) siendo E= C/número de genes del genoma, luego el valor de R seria equivalente al valor Observado(O) entre el valor esperado(E), donde a mayor R el valor de enriquecimiento es mayor, donde indica que hay más de lo esperado al azar, siendo una "función enriquecida" donde el valor alto de enriquecimiento está asociado a un valor de p bastante bajo.
FUENTE: https://www.webgestalt.org/2013/htdocs/final_KEGG_file_1567526521.html
39.5. STRING DATABASE (interacciones proteína-proteína)
¿En que contexto o problemática se aplican?
Para este estudio, se usa una base de datos que muestra las interaciones proteína-proteina, donde estas interacciones no necesariamente son físicas, son más bien relaciones. En el string se observa los Score, siendo una evidencia experimental donde hay una reacción real directa de una proteína que se une a otra, y lo activa, inhibe o modula su función, donde las interacciones proteína-proteína son anotados con score que van del valor de 0 a 1. El valor de 1 indica la máxima cantidad de relaciones posibles entre dos proteínas. El String Database, luego de añadir la lista de genes en Excel en archivo TXT, donde nos muestra una gráfica de interacciones en la cual los nodos de la red representan las proteínas, y los bordes representan las interacciones proteína-proteína, por ejemplo en coocurrencia esos dos genes están mencionado en la publicación científica de forma incidental, cada uno de los colores tiene un significado, donde el grosor de la línea, indica el nivel de la fuerza de los datos que apoya la interacción, en cambio, si se aumenta la confidencia, las interacciones son muy robustas y si bajo la confidencia, las interacciones van a ser más débiles y directas.
39.6. Data mining-GEO (Gene Expression Omnibus)
https://www.ncbi.nlm.nih.gov/geo/
¿En qué contexto o problemática se aplican?
Es una base de datos en
la que los investigadores depositan sus datos experimentales de genómica de
microarreglos, Transcriptómica, proteómica de secuenciación, esos datos son públicos,
de libre acceso, se puede hacer DATA MINING-MINERIA DE DATOS, sobre datos que
están publicados en el GEO, no es la única, pero es una de las más usadas. En
el GEO cada experimento es depositado en el fondo de su totalidad, tanto los
datos crudos, como los procesados deben ser depositados, subidos a esa
plataforma y eventualmente como usuario se puede descargar los datos crudos no
procesados, buscando códigos de experimentos que se suben en el conjunto de
muestras, analizando con el mismo microarrays de tal forma que un código por
ejemplo GSE=37493; GSE=47185 siendo una especie de meta-análisis, los clasifica
por diferentes tipos de muestras, el cual nos permite distinguir diferentes
subtipos de enfermedad, con el código accedo al experimento, el cual tiene título
en experiment type, esto va asociado a uno o más paper siendo lo más importante
las plataformas que fueron usadas, así como el affimetrix.
40.- SOFTWARE QUE USAMOS PARA NUESTROS ESTUDIOS EN "FISIOLOGÍA Y BIOTECNOLOGÍA MOLECULAR VEGETAL"
40.1. Análisis de vías metabólicas de Arabidopsis thaliana, AraCyc: https://www.arabidopsis.org/
40.2. Búsqueda de posibles ortológos en otras especies vegetales en Base de datos de PlantGDB, utilizando programa bioinformático BLAST: https://www.plantgdb.org/
40.3. Alineamiento multiple: www.ebi.ac.uk/Tools/msa/clustalo/
40.4. Análisis de expresión génica en Arabidopsis y otras especies, usando datos de microarreglos/RNAseq: https://bar.utoronto.ca/efp2/Arabidopsis/Arabidopsis_eFPBrowser2.html
https://www.plantgdb.org/cgi-bin/blast/PlantGDBblast
41. SOFTWARE QUE USAMOS PARA NUESTROS ESTUDIOS EN "RITMOS CIRCADIANOS"
41.1. Circadian Expression Profiles Data base: https://circadb.hogeneschlab.org/about
42. Análisis de datos bioinformáticos para metagenomas y amplicones usando R (del Dr. Eduardo Castro Nallar; Universidad Andrés Bello, Chile) ISME Latin America 2019
El curso cuenta con seis unidades, cada una un el objetivo de conocer y practicar una habilidad de análisis diferente de datos de secuenciación de amplicones o metagenomas usando el software R. Sino tienes experiencia trabajando en R, te recomendamos comenzar con la unidad 1: "Introducción a R: Manipulación de datos y visualización".
Por favor, revisa la lista de unidades disponibles a continuación y haz clic sobre la que quieras practicar para dirigirte al tutorial.
- Introducción a R: Manipulación de datos y visualización
- Análisis de secuencias de 16S con DADA2
- Introducción a phyloseq y a análisis de diversidad
- Búsqueda de genes de interés en datos de metagenómica shotgun
- Visualización y curación de genomas ensamblados desde metagenomas (MAGs)
- Redes de co-ocurrencia de microorganismos
Nota: Para los días del curso suponemos que los participantes han instalado R y RStudio y los paquetes de R necesarios, siguiendo la guía de requerimientos enviada una semana antes. De lo contrario, puedes hacerlo siguiendo los pasos en el siguiente link:
Antes de comenzar: Instalación de R, RStudio y paquetes
Este curso es diseñado e impartido por el grupo de Dr. Eduardo Castro-Nallar (eduardo.castro@unab.cl):Dr. Florence Gutzwiller (florence.gutzwiller@gmail.com) y M.Sc. Katterinne N. Mendez (mendez.katterinne@gmail.com)
Antes de comenzar
El trabajo práctico del curso se basa en el análisis de datos y visualización en el lenguaje y entorno computacional para análisis estadísticos y gráficos R, usando RStudio como plataforma de trabajo.
Por favor, sigue los pasos para instalar R, RStudio, y los paquetes de R necesarios para los análisis que practicaremos en el curso.
R y RStudio
El término R se refiere tanto al lenguaje de programación como al software que interpreta los scripts escritos usándolo. R es un software de libre acceso para computar análisis estadísticos y construir gráficos representativos. RStudio es la forma más popular y reconocida para escribir R scripts e interactuar con el software, funciona como interfaz gráfica de R, para hacer su uso más fácil e interactivo. Ambos están disponibles para Linux, MacOS y Windows.
¿Ya tienes R y/o RStudio instalados?
- Abre RStudio, haz clic en "Help" -> "Check for updates". Si hay una nueva versión disponible, cierra RStudio y descarga e instala la última versión https://www.rstudio.com/products/rstudio/download/#download
- Para saber qué versión de R estás usando, simplemente inicia RStudio y lo primero que verás en la consola es la versión de R, o bien, puedes escribir sessionInfo() en la consola de R o RStudio. Ve a la página de CRAN https://cran.r-project.org/ para ver si hay una versión más reciente de R disponible, si es así descarga e instala la última versión.
Instala R y RStudio
Necesitas instalar R antes de instalar RStudio.
- Dirígete a la página de CRAN y descarga e instala R, según corresponda para tu sistema operativo.
- Dirígete a la página de descarga de RStudio y descarga e instala RStudio, según corresponda para tu sistema operativo. Una vez instalado, abre RStudio y asegúrate de que no aparece algún mensaje de error en la consola.
- ¿Tienes algún problema o duda? Dale un vistazo a la página https://cran.r-project.org/doc/FAQ/R-FAQ.html
- ¿MacOS? Te recomendamos instalar https://developer.apple.com/xcode/ y https://www.xquartz.org/
Instalar y cargar paquetes en R
Los repositorios de paquetes de R que vamos utilizar son: https://cran.r-project.org/ https://www.bioconductor.org/ y https://github.com/- Sigue los pasos a continuación para instalar y cargar la lista de paquetes del curso.
- Definir lista de paquetes según repositorio cran_packages <- c("devtools", "igraph", "qgraph", "RColorBrewer", "tidyverse", "ggplots", "gplots", "network", "sna", "seqinr", "plyr", "qtl", "magrittr", "grid", "gridExtra", "dplyr", "pheatmap", "xtable", "kableExtra", "remotes", "Rtsne", "vegan") bioc_packages <- c("phyloseq", "microbiome", "genefilter", "Rbowtie", "dada2", "DECIPHER", "phangorn", "ggpubr", "BiocInstaller","DESeq2", "genefilter", "philr", "GenomeInfoDb", "microbiome") git_source <- c("zdk123/SpiecEasi", "hallucigenia-sparsa/seqtime", "briatte/ggnet", "twbattaglia/btools", "gmteunisse/Fantaxtic", "MadsAlbertsen/ampvis2", "opisthokonta/tsnemicrobiota", "") git_packages <- c("SpiecEasi", "seqtime", "ggnet", "btools", "fantaxtic", "ampvis2", "tsnemicrobiota")
- Instalar paquetes según reporitorio
- Cargar paquetes y confirmar que fueron todos instalados correctamente
Finalmente, en la consola se muestra cada paquete acompañado de la palabra TRUE o FALSE, si todos los paquetes marcan TRUE significa que fueron cargados exitosamente.
Una vez instalados los paquetes en R, solo es necesario cargar aquellos necesarios a la sessión actual de R usando la función library()
# Cargar paquetes library(tidyverse) library(phyloseq)42.1. Introducción R, manipulación y visualización en R
42.1..1 Acerca del curso
42.1.1.1. Introducción a R
1.1 ¿Por qué R?
Tus análisis serán una serie de comandos escritos (R script), lo que es muy conveniente porque:
- Conlleva a tener el control y completo entendimiento de qué estás haciendo (adiós a la caja negra en tus análisis in silico).
- ¡Reproducibilidad! Si necesitas repetir el análisis, agregar más datos o corregir un error, sólo necesitas correr tu script nuevamente y tus tests estadísticos y gráficos se actualizarán automáticamente.
- Tus análisis son claros y transparentes, cualquier colega o tú en el futuro pueden leerlo para encontrar errores y/o hacer mejoras.
- Puedes comentar tu script. Agregar comentarios, explicaciones o relatos en tu código facilitará entender, paso a paso, el por qué de tus análisis a tus colegas, e incluso a ti mísmo en el futuro.
Otras importantes ventajas de usar R son:
- ¡Escalabilidad! R está diseñado para el análisis de datos. Las habilidades que vayas aprendiendo en R se pueden escalar fácilmente con el tamaño de tus datos (set de datos con cientos a miles, incluso millones de líneas).
- ¡Gráficos de alta calidad! R tiene innumerables funcionalidades para todo tipo de gráficas a tu disposición para lograr una efectiva visualización de tus resultados. Visita https://www.r-graph-gallery.com/all-graphs para ver ejemplos de lo que puedes llegar a hacer.
- ¡Amplia documentación! R cuenta con una basta documentación y la web está minada de tutoriales, sólo debes buscar. Por ejemplo https://www.cookbook-r.com/ que provee de soluciones a tareas y problemas básicos en análisis de datos.
- Aprender R puede ser difícil y tomar tiempo en un principio, pero ¡no te preocupes, no estás sólo! R cuenta con una amplia comunidad de usuarios dispuestos a ayudar a través de mailing-lists y websites como https://stackoverflow.com/ y https://community.rstudio.com/. Sin embargo, lo más probable es que tus dudas ya se encuentren resueltas en la web, asi que primero haz una búsqueda en Google usando palabras clave o copia y pega en el buscador el mensaje de error que te aparezca en la consola de R. Consejo: haz tus búsquedas y consultas en inglés, tendrás acceso a más y mejor información.
1.2 Conociendo RStudio
¡Todo lo que necesitas en una ventana! Como puedes ver en la imagen a continuación, RStudio se divide en 4 paneles principales:
- Panel superior izquierdo: editor de texto para escribir comandos/instrucciónes en R (R script), aquí puedes agregar comentarios.
- Panel inferior izquierdo: consola de R, donde se ejecutan las líneas de comando en tu script.
- Panel superior derecho: aquí puedes visualizar los datos presentes en la memoria de R.
- Panel inferior derecho: aquí encuentras 5 pestañas Files/Plots/Packages/Help/Viewer en las que puedes navegar por tus archivos, visualizar tus gráficos en tiempo real, administrar paquetes de R, y pedir ayuda.

RStudio interfaz
Básicamente, lo que haremos es escribir instrucciones (a lo que llamamos comandos) en el lenguaje de programación R, y luego le indicaremos al computador que ejecute la instrucción (a lo que llamamos ejecutar o correr). En RStudio puedes escribir los comandos en el editor de texto y luego ejecutarlos, una línea de código a la vez, presionando Ctrl + Enter (cmd + return en Mac). Cuando ejecutas comandos, es importante poner atención a la consola. Si hubo algún problema con la ejecución de la instrucción, aparecerá un mensaje de error en la consola. Cuando el computador finaliza la instrucción, un nuevo > aparece en la consola indicando que a finalizado de ejecutar el último comando y está listo para correr el siguiente.
1.3 Set de datos para trabajar
Para ésta sección vamos a trabajar con una tabla de datos que contiene información acerca de índices de diversidad de microorganismos presentes en 87 muestras de piel de 3 especies de ballena recolectadas en 3 zonas de Chile (Megaptera novaeangliae, Balaenoptera musculus, Balaenoptera physalus; Estrecho Magallanes, Chiloé, Reserva Nacional Pinguino de Humboldt).
Descarga la tabla de datos IR_table1.csv https://www.dropbox.com/s/htbjbqsdrcinrcr/IR_table1.csv?dl=0
Usa la función read.table() para leer o cargar la tabla de datos a la memoria de R. La forma de cargar nuevos datos a la memoria de R, es asignar dicho set de datos a un objeto usando <- (objeto <- datos).
data <- read.table(file = "~/Dropbox/CastroLab_database/workshops_data/IR_table1.csv", sep = ",", header = TRUE) # usa la ruta correspondiente en tu computadora hasta el archivo 'IR_table1.csv' # ahora los datos se encuentran guardados en el objeto "data"
file = "", sep = "" y header = TRUE/FALSE son argumentos de la función read.table. Cada vez que llamamos una función debemos usar argumentos para indicar datos de entrada y/o preferencias:
- file = "~/Dropbox/CastroLab_database/workshops_data/IR_table1.csv" para indicar archivo del cual queremos copiar datos y cargarlos a la memoria de R.
- sep = "," para indicar el separador de columnas de la tabla.
- header = TRUE para indicar que la tabla "IR_table1.csv" sí ("TRUE") contiene headers o títulos de columna, de lo contrario usaríamos "FALSE".
Puedes conocer la descripción de cualquier función y sus argumentos escribiendo "?" + "nombre de la función" en la consola de R (e.g., ?read.table).
Cuando cargamos una tabla de datos usando la función read.table(), ésta pasa a conformar lo que se conoce como un data frame en la memoria de R. Un data frame es una representación de los datos en forma de tabla, donde las columnas son vectores, todos del mismo largo (igual número de filas). Un vector es el tipo de dato más básico en R, está compuesto por una serie de valores los que pueden ser números o caracteres.
- Usa la función View() para ver el contenido del objeto data.
View(data)
Prueba las siguientes funciones para inspeccionar nuestro nuevo data frame "data": dim(data) # muestra número de filas y columnas, respectivamente
## [1] 87 30
nrow(data) # muestra número de filas
## [1] 87
ncol(data) # muestra número de columnas
## [1] 30
head(data) # muestra las primeras 6 filas
## sample_ID geo_loc_name species observed ## 1 SRR6442697 Estrecho de Magallanes Megaptera novaeangliae 31
## 2 SRR6442698 Estrecho de Magallanes Megaptera novaeangliae 33
## 3 SRR6442699 Estrecho de Magallanes Megaptera novaeangliae 43
## 4 SRR6442700 Estrecho de Magallanes Megaptera novaeangliae 29
## 5 SRR6442701 Estrecho de Magallanes Megaptera novaeangliae 26
## 6 SRR6442702 Estrecho de Magallanes Megaptera novaeangliae 21
## shannon richness_0 richness_20 richness_50 richness_80
## 1 2.121629 31 31 31 31
## 2 1.499137 33 33 33 33
## 3 2.249373 43 43 43 43
## 4 1.306574 29 29 29 29
## 5 1.077438 26 26 26 26
## 6 1.131204 21 21 21 21
## diversities_inverse_simpson diversities_gini_simpson diversities_shannon
## 1 6.112104 0.8363902 2.121629
## 2 2.505873 0.6009375 1.499137
##3 7.388570 0.8646558 2.249373
## 4 2.644111 0.6218011 1.306574
## 5 1.873331 0.4661914 1.077438
## 6 2.740500 0.6351030 1.131204
## diversities_fisher diversities_coverage evenness_camargo evenness_pielou
## 1 3.907415 3 0.02929889 0.6178324
## 2 4.844428 1 0.01815948 0.4287528
## 3 5.368153 3 0.03391424 0.5980465
## 4 3.094775 1 0.01389960 0.3880188
## 5 2.956294 1 0.01143026 0.3306954
## 6 2.010698 2 0.01179407 0.3715539
## evenness_simpson evenness_evar evenness_bulla dominance_dbp
## 1 0.028296778 0.08554199 0.06038316 0.3022210
## 2 0.011601264 0.11572176 0.05579809 0.6138276
## 3 0.034206345 0.07397473 0.05992808 0.2424974
## 4 0.012241256 0.05943919 0.04011332 0.5250103
## 5 0.008672827 0.08300059 0.04374973 0.7143736
## 6 0.012687499 0.05585996 0.02383196 0.4903917
## dominance_dmn dominance_absolute dominance_relative dominance_simpson
## 1 0.4948605 3293 0.3022210 0.1636098
## 2 0.7063907 2699 0.6138276 0.3990625
## 3 0.4386486 3919 0.2424974 0.1353442
## 4 0.8384308 19071 0.5250103 0.3781989
## 5 0.8534960 13936 0.7143736 0.5338086
## 6 0.7767577 33864 0.4903917 0.3648970
## dominance_core_abundance dominance_gini rarity_log_modulo_skewness
## 1 0.5595631 0.9707011 2.055226
## 2 0.8749147 0.9818405 2.060118
## 3 0.5033723 0.9660858 2.059857
## 4 0.9925120 0.9861004 2.057839
## 5 0.9824175 0.9885697 2.056901
## 6 0.9981030 0.9882059 2.049704
## rarity_low_abundance rarity_noncore_abundance rarity_rare_abundance
## 1 0.009177680 0.076174743 0.076174743
## 2 0.007505117 0.024789629 0.024789629
## 3 0.005692717 0.282965163 0.282965163
## 4 0.002890571 0.000220234 0.000220234
## 5 0.004459709 0.001127742 0.001127742
## 6 0.003301716 0.000086900 0.000086900
## 82 SRR6442787 Estrecho de Magallanes Megaptera novaeangliae 34
## 83 SRR6442788 Estrecho de Magallanes Megaptera novaeangliae 49
## 84 SRR6442789 Estrecho de Magallanes Megaptera novaeangliae 28
## 85 SRR6442790 Estrecho de Magallanes Megaptera novaeangliae 27
## 86 SRR6442792 Estrecho de Magallanes Megaptera novaeangliae 24
## 87 SRR6442794 Estrecho de Magallanes Megaptera novaeangliae 22
## shannon richness_0 richness_20 richness_50 richness_80
## 82 1.2165375 34 34 34 34
## 83 1.4508829 49 49 49 49
## 84 0.8779605 28 28 28 28
## 85 1.2337859 27 27 27 27
## 86 1.6592270 24 24 24 24
## 87 1.0682126 22 22 22 22
## diversities_inverse_simpson diversities_gini_simpson
## 82 2.370590 0.5781641
## 83 2.723141 0.6327769
## 84 2.132005 0.5309578
## 85 2.365953 0.5773373
## 86 3.274365 0.6945973
## 87 2.195027 0.5444248
## diversities_shannon diversities_fisher diversities_coverage
## 82 1.2165375 3.789528 1
## 83 1.4508829 5.808398 1
## 84 0.8779605 3.261216 1
## 85 1.2337859 3.494975 1
## 86 1.6592270 3.350736 1
## 87 1.0682126 2.388742 1
## evenness_camargo evenness_pielou evenness_simpson evenness_evar
## 82 0.01240594 0.3449839 0.010974954 0.07523218
## 83 0.01636409 0.3728032 0.012607132 0.10265990
## 84 0.01006692 0.2634774 0.009870391 0.10082495
## 85 0.01243090 0.3743468 0.010953485 0.09603370
## 86 0.01921725 0.5220890 0.015159097 0.10629033
## 87 0.01062587 0.3455833 0.010162163 0.06442340
## evenness_bulla dominance_dbp dominance_dmn dominance_absolute
## 82 0.02951427 0.6173890 0.7687722 18434
## 83 0.05934702 0.5651963 0.7234154 15132
## 84 0.02545193 0.5161512 0.9659794 9012
## 85 0.03539774 0.6129155 0.8023506 4850
## 86 0.05534884 0.5129630 0.6608796 2216
## 87 0.03291487 0.6099778 0.8939806 14562
## dominance_relative dominance_simpson dominance_core_abundance
## 82 0.6173890 0.4218359 0.9939380
## 83 0.5651963 0.3672231 0.8883203
## 84 0.5161512 0.4690422 0.9822451
## 85 0.6129155 0.4226627 0.9962088
## 86 0.5129630 0.3054027 0.8837963
## 87 0.6099778 0.4555752 0.9965652
## dominance_gini rarity_log_modulo_skewness rarity_low_abundance
## 82 0.9875941 2.052933 0.003181727
## 83 0.9836359 2.057605 0.015164531
## 84 0.9899331 2.057562 0.006815578
## 85 0.9875691 2.059455 0.007456085
## 86 0.9807828 2.058916 0.004166667
## 87 0.9893741 2.054947 0.002303858
## rarity_noncore_abundance rarity_rare_abundance
## 82 0.000837297 0.000837297
## 83 0.014044000 0.014044000
## 84 0.014604811 0.014604811
## 85 0.000884620 0.000884620
## 86 0.001157407 0.001157407
## 87 0.000376995 0.000376995
summary(data) # calcula estadísticas básicas para cada columna
## sample_ID geo_loc_name
## SRR6442697: 1 Chiloe :28
## SRR6442698: 1 Estrecho de Magallanes :41
## SRR6442699: 1 Reserva Nacional Pinguino de Humboldt:18
## SRR6442700: 1
## SRR6442701: 1
## SRR6442702: 1
## (Other) :81
## species observed shannon
## Balaenoptera musculus :26 Min. :13.0 Min. :0.1675
## Balaenoptera physalus : 7 1st Qu.:28.0 1st Qu.:1.1493
## Megaptera novaeangliae:54 Median :36.0 Median :1.4509
## Mean :41.3 Mean :1.4610
## 3rd Qu.:53.0 3rd Qu.:1.7976
## Max. :84.0 Max. :3.1401
## ## richness_0 richness_20 richness_50 richness_80
## Min. :13.0 Min. :13.0 Min. :13.0 Min. :13.0
## 1st Qu.:28.0 1st Qu.:28.0 1st Qu.:28.0 1st Qu.:28.0
## Median :36.0 Median :36.0 Median :36.0 Median :36.0
## Mean :41.3 Mean :41.3 Mean :41.3 Mean :41.3
## 3rd Qu.:53.0 3rd Qu.:53.0 3rd Qu.:53.0 3rd Qu.:53.0
## Max. :84.0 Max. :84.0 Max. :84.0 Max. :84.0
## ## diversities_inverse_simpson diversities_gini_simpson diversities_shannon
## Min. : 1.052 Min. :0.04979 Min. :0.1675
## 1st Qu.: 2.164 1st Qu.:0.53769 1st Qu.:1.1493
## Median : 2.814 Median :0.64461 Median :1.4509
## Mean : 3.478 Mean :0.63208 Mean :1.4610
## 3rd Qu.: 4.137 3rd Qu.:0.75821 3rd Qu.:1.7976
## Max. :14.576 Max. :0.93139 Max. :3.1401
## ## diversities_fisher diversities_coverage evenness_camargo
## Min. : 1.620 Min. :1.000 Min. :0.005441
## 1st Qu.: 3.286 1st Qu.:1.000 1st Qu.:0.012368
## Median : 4.056 Median :2.000 Median :0.016613
## Mean : 5.018 Mean :1.701 Mean :0.018888
## 3rd Qu.: 6.263 3rd Qu.:2.000 3rd Qu.:0.023079
## Max. :13.163 Max. :5.000 Max. :0.083105
## ## evenness_pielou evenness_simpson evenness_evar evenness_bulla
## Min. :0.04711 Min. :0.004872 Min. :0.05064 Min. :0.01145
## 1st Qu.:0.32381 1st Qu.:0.010016 1st Qu.:0.06995 1st Qu.:0.03536
## Median :0.38802 Median :0.013027 Median :0.09603 Median :0.04923
## Mean :0.40233 Mean :0.016100 Mean :0.10164 Mean :0.05415
## 3rd Qu.:0.49853 3rd Qu.:0.019151 3rd Qu.:0.12267 3rd Qu.:0.06665
## Max. :0.74418 Max. :0.067482 Max. :0.25815 Max. :0.19846
## ## dominance_dbp dominance_dmn dominance_absolute dominance_relative
## Min. :0.1358 Min. :0.2602 Min. : 382 Min. :0.1358
## 1st Qu.:0.3772 1st Qu.:0.6092 1st Qu.: 4856 1st Qu.:0.3772
## Median :0.4841 Median :0.7482 Median :13936 Median :0.4841
## Mean :0.5042 Mean :0.7221 Mean :16988 Mean :0.5042
## 3rd Qu.:0.6167 3rd Qu.:0.8478 3rd Qu.:20533 3rd Qu.:0.6167
## Max. :0.9747 Max. :0.9932 Max. :80901 Max. :0.9747
## ## dominance_simpson dominance_core_abundance dominance_gini
## Min. :0.06861 Min. :0.0063 Min. :0.9169
## 1st Qu.:0.24179 1st Qu.:0.4664 1st Qu.:0.9769
## Median :0.35539 Median :0.9189 Median :0.9834
## Mean :0.36792 Mean :0.7313 Mean :0.9811
## 3rd Qu.:0.46231 3rd Qu.:0.9736 3rd Qu.:0.9876
## Max. :0.95021 Max. :0.9988 Max. :0.9946
## ## rarity_log_modulo_skewness rarity_low_abundance rarity_noncore_abundance
## Min. :1.976 Min. :0.001072 Min. :0.0000869
## 1st Qu.:2.054 1st Qu.:0.004414 1st Qu.:0.0017489
## Median :2.058 Median :0.007645 Median :0.0062500
## Mean :2.053 Mean :0.010067 Mean :0.0630320
## 3rd Qu.:2.060 3rd Qu.:0.013359 3rd Qu.:0.0252855
## Max. :2.061 Max. :0.039322 Max. :0.9751045
## ## rarity_rare_abundance
## Min. :0.0000869
## 1st Qu.:0.0017489
## Median :0.0062500
## Mean :0.0630320
## 3rd Qu.:0.0252855
## Max. :0.9751045
##
1.4 Extraer información de tablas
Nuestra tabla de datos de estudio (data) consta de filas y columnas (2 dimensiones), si queremos extraer algunos datos de interés, debemos especificar las "coordinadas" de los datos que queremos obtener. Primero el número(s) de fila, seguido por el número(s) de columna. Existen diferentes formas de especificar coordenadas, que nos llevaran a obtener datos de diferente clase o tipo.
- Primer elemento en la primera columna del data frame, como vector:
data[1, 1]
- Primer elemento en la sexta columna, como vector:
data[1, 6]
- Primera columna del data frame, como vector:
data[, 1]
- Primera columna del data frame, como data frame:
data[1]
- Primeros tres elementos en la séptima columna, como vector:
data[1:3, 7] # filas 1 a 3 de la columna 7
- La tercera fila del data frame, como data frame:
data[3, ]
- Equivalente a la función head():
data[1:6, ]
- También puedes excluir datos usando el símbolo - ("todo menos..."):
data[, -1] # todo excepto la primera columna data[-c(7:87), ] # equivalente a head(data)
La función c() se utiliza para indicar una serie de valores o asignar una serie de valores a un vector (e.g., peso_kg <- c(60,55,64,80,74)).
- También puedes extraer datos usando el nombre de las columnas o headers:
data["species"] # resultado como data.frame data[, "species"] # resultado como vector data[["species"]] # resultado como vector data$species # resultado como vector
Puedes revisar los headers disponibles en tu data frame usando las funciones colnames(data) o View(data).
RStudio cuenta con una muy útil función de autocompletado, presiona tab (tabulador) para obtener nombres completos y correctos de funciones, columnas (headers), etc.
1.5 Factores
- La función str() muestra la estructura de un objeto e información acerca de la clase y contenido de cada columna:
Como podrás ver en el output de str(data) que las columnas sample_ID, geo_loc_name y species son de una clase llamada Factor. Factores representan datos categóricos. Son guardados en la memoria de R como números enteros (integers), los que pueden estar ordenados o desordenados.
Los factores contienen un set de valores pre-definidos, conocidos como levels. Por defecto, R ordena los levels en orden alfabético. Por ejemplo, en nuestro objeto data la columna species es un Factor con 3 levels:
levels(data$species)## [1] "Balaenoptera musculus" "Balaenoptera physalus" ## [3] "Megaptera novaeangliae"nlevels(data$species)## [1] 3R asigna 1 al level "Balaenoptera musculus", 2 al level "Balaenoptera physalus" y 3 al level "Megaptera novaeangliae".
Algunas veces, el orden de los factores no importa, pero otras veces vamos a requerir especificar el orden porque es importante para el análisis o visualización de los datos. Una forma de re-ordenar los levels del factor species es:
levels(data$species) # orden actual## [1] "Balaenoptera musculus" "Balaenoptera physalus" ## [3] "Megaptera novaeangliae"data$species <- factor(data$species, levels = c("Megaptera novaeangliae", "Balaenoptera musculus", "Balaenoptera physalus")) levels(data$species) # después de re-ordenar## [1] "Megaptera novaeangliae" "Balaenoptera musculus" ## [3] "Balaenoptera physalus"1.5.1 Transformar factores
- Para transformar un factor a un vector de caracteres, puedes usar la función as.character():
- Transformar o convertir factores cuyos niveles son números (e.g., años) a un vector numérico es un poco más complejo. La función as.numeric() muestra los números enteros asignados a cada level, no los niveles en si. Una manera de evitarlo es convertir los factores a caracteres, y luego a números:
1.5.2 Renombrar factores
¿Necesitas renombrar algún elemento en tus datos? Supongamos que queremos cambiar el nombre de la especie "Megaptera novaeangliae" por el nombre común "ballena jorobada".
species <- data$species # copiamos la columna "species" en un objeto aparte para no alterar nuestro set de datos original head(species)## [1] Megaptera novaeangliae Megaptera novaeangliae Megaptera novaeangliae ## [4] Megaptera novaeangliae Megaptera novaeangliae Megaptera novaeangliae ## 3 Levels: Megaptera novaeangliae ... Balaenoptera physaluslevels(species) # identifica la posición del level que quieres renombrar (1)## [1] "Megaptera novaeangliae" "Balaenoptera musculus" ## [3] "Balaenoptera physalus"levels(species)[1] <- "ballena jorobada" levels(species)## [1] "ballena jorobada" "Balaenoptera musculus" "Balaenoptera physalus"head(species)## [1] ballena jorobada ballena jorobada ballena jorobada ballena jorobada ## [5] ballena jorobada ballena jorobada ## 3 Levels: ballena jorobada ... Balaenoptera physalus# también puedes hacerlo para los otros dos levels levels(species)[2] <- "ballena azul" levels(species)[3] <- "ballena de aleta" levels(species)## [1] "ballena jorobada" "ballena azul" "ballena de aleta"1.5.3 Argumento stringsAsFactors
Por defecto, al importar un data frame en R, las columnas que contienen caracteres (i.e. texto) son convertidas en factores. Dependiendo de qué queramos hacer con los datos, en algún caso podríamos necesitar que la columna se mantenga como caracter. Para ésto, la función read.table() tiene disponible el argumento stringsAsFactors que puede ser definido como "FALSE" (stringsAsFactors = FALSE).
- Compara la diferencia entre la tabla de datos de estudio leída como factor vs. caracter:
.
2. Manipulación de datos en R
Las funciones que hemos estado usando hasta ahora, como read.table y str(), vienen incorporadas en R. Pero existen incontables funciones en R a las que puedes acceder instalando lo que se conoce como paquetes (packages). Antes de usar un paquete por primera vez, primero debes instalarlo y luego debes importarlo en cada sesión de R en la que quieras usarlo.
Hay dos formas de instalar paquetes en R, a través de la consola y a través de la opción Install en la pestaña Packages de RStudio:
- Para instalar e importar el paquete tidyverse usando la consola, escribe:
- Para instalar e importar el paquete tidyverse usando RStudio, dirígete a la pestaña Packages -> Install -> escribe el nombre del paquete y haz clic en "Install". Una vez instalado, para importarlo sólo debes identificarlo y seleccionarlo en la lista de paquetes.
Instalación de paquetes en RStudio
tidyverse es un paquete que incluye la instalación varios paquetes ´útiles para la manipulaci´ón y an´álisis de datos, tales como dplyr, tidyr, ggplot2, etc. Anteriormente practicamos como extraer información de tablas usando [ ], a continuación vamos a utilizar los paquetes dplyr y tidyr para manipular nuestra tabla de datos más fácil, rápido y con más funcionalidades.
- Vamos a leer nuevamente nuestra tabla de datos, ésta vez usando la función read_csv() del paquete tidyverse:
data <- read_csv("~/Dropbox/CastroLab_database/workshops_data/IR_table1.csv")
## Parsed with column specification:
## cols( ## .default = col_double(),
## sample_ID = col_character(),
## geo_loc_name = col_character(),
## species = col_character()
## )
## See spec(...) for full column specifications.
# Usa la función str() para inspeccionar los datos
str(data)
## Classes 'spec_tbl_df', 'tbl_df', 'tbl' and 'data.frame': 87 obs. of 30 variables:
## $ sample_ID : chr "SRR6442697" "SRR6442698" "SRR6442699" "SRR6442700" ...
## $ geo_loc_name : chr "Estrecho de Magallanes" "Estrecho de Magallanes" "Estrecho de Magallanes" "Estrecho de Magallanes" ...
## $ species : chr "Megaptera novaeangliae" "Megaptera novaeangliae" "Megaptera novaeangliae" "Megaptera novaeangliae" ...
## $ observed : num 31 33 43 29 26 21 37 32 28 30 ...
## $ shannon : num 2.12 1.5 2.25 1.31 1.08 ...
## $ richness_0 : num 31 33 43 29 26 21 37 32 28 30 ...
## $ richness_20 : num 31 33 43 29 26 21 37 32 28 30 ...
## $ richness_50 : num 31 33 43 29 26 21 37 32 28 30 ...
## $ richness_80 : num 31 33 43 29 26 21 37 32 28 30 ...
## $ diversities_inverse_simpson: num 6.11 2.51 7.39 2.64 1.87 ...
## $ diversities_gini_simpson : num 0.836 0.601 0.865 0.622 0.466 ...
## $ diversities_shannon : num 2.12 1.5 2.25 1.31 1.08 ...
## $ diversities_fisher : num 3.91 4.84 5.37 3.09 2.96 ...
## $ diversities_coverage : num 3 1 3 1 1 2 2 1 2 1 ...
## $ evenness_camargo : num 0.0293 0.0182 0.0339 0.0139 0.0114 ...
## $ evenness_pielou : num 0.618 0.429 0.598 0.388 0.331 ...
## $ evenness_simpson : num 0.0283 0.0116 0.03421 0.01224 0.00867 ...
## $ evenness_evar : num 0.0855 0.1157 0.074 0.0594 0.083 ...
## $ evenness_bulla : num 0.0604 0.0558 0.0599 0.0401 0.0437 ...
## $ dominance_dbp : num 0.302 0.614 0.242 0.525 0.714 ...
## $ dominance_dmn : num 0.495 0.706 0.439 0.838 0.853 ...
## $ dominance_absolute : num 3293 2699 3919 19071 13936 ...
## $ dominance_relative : num 0.302 0.614 0.242 0.525 0.714 ...
## $ dominance_simpson : num 0.164 0.399 0.135 0.378 0.534 ...
## $ dominance_core_abundance : num 0.56 0.875 0.503 0.993 0.982 ...
## $ dominance_gini : num 0.971 0.982 0.966 0.986 0.989 ...
## $ rarity_log_modulo_skewness : num 2.06 2.06 2.06 2.06 2.06 ...
## $ rarity_low_abundance : num 0.00918 0.00751 0.00569 0.00289 0.00446 ...
## $ rarity_noncore_abundance : num 0.07617 0.02479 0.28297 0.00022 0.00113 ...
## $ rarity_rare_abundance : num 0.07617 0.02479 0.28297 0.00022 0.00113 ...
## - attr(*, "spec")=
## .. cols(
## .. sample_ID = col_character(),
## .. geo_loc_name = col_character(),
## .. species = col_character(),
## .. observed = col_double(),
## .. shannon = col_double(),
## .. richness_0 = col_double(),
## .. richness_20 = col_double(),
## .. richness_50 = col_double(),
## .. richness_80 = col_double(),
## .. diversities_inverse_simpson = col_double(),
## .. diversities_gini_simpson = col_double(),
## .. diversities_shannon = col_double(),
## .. diversities_fisher = col_double(),
## .. diversities_coverage = col_double(),
## .. evenness_camargo = col_double(),
## .. evenness_pielou = col_double(),
## .. evenness_simpson = col_double(),
## .. evenness_evar = col_double(),
## .. evenness_bulla = col_double(),
## .. dominance_dbp = col_double(),
## .. dominance_dmn = col_double(),
## .. dominance_absolute = col_double(),
## .. dominance_relative = col_double(),
## .. dominance_simpson = col_double(),
## .. dominance_core_abundance = col_double(),
## .. dominance_gini = col_double(),
## .. rarity_log_modulo_skewness = col_double(),
## .. rarity_low_abundance = col_double(),
## .. rarity_noncore_abundance = col_double(),
## .. rarity_rare_abundance = col_double()
## .. )
Como podrás notar, la función read_csv() hace algunos cambios al cargar la tabla con respecto a lo que revisamos anteriormente usando read.table(). Las diferencias son:
- Al leer la tabla, muestra un resumen del tipo de dato de cada columna, y sólo muestra las primeras filas y tantas columnas como se puedan visualizar en la pantalla.
- Las columnas de clase character (caracteres) no son convertidas en factores.
A continuación vamos a aprender algunas de las funciones más comunes de dplyr:
- select(): extraer columnas.
- filter(): extraer filas según condiciones.
- mutate(): crear nuevas columnas usando la información de otras columnas.
- group_by() y summarize(): cálcula estadísticas en datos agrupados.
- arrange(): ordena resultados.
- count(): conteo de datos.
2.1 Seleccionar columnas y filtrar filas
- Para seleccionar columnas de una tabla de datos o data frame, vamos a usar la función select(). Luego, para seleccionar filas de acuerdo a cierto criterio (filtrar), vamos a usar la función filter().
select(data, sample_ID, geo_loc_name, species, observed) # los argumentos son, primero el objeto que contiene el data frame, y luego los títulos de las columnas a extraer
## # A tibble: 87 x 4
## sample_ID geo_loc_name species observed
## <chr> <chr> <chr> <dbl>
## 1 SRR6442697 Estrecho de Magallanes Megaptera novaeangliae 31
## 2 SRR6442698 Estrecho de Magallanes Megaptera novaeangliae 33
## 3 SRR6442699 Estrecho de Magallanes Megaptera novaeangliae 43
## 4 SRR6442700 Estrecho de Magallanes Megaptera novaeangliae 29
## 5 SRR6442701 Estrecho de Magallanes Megaptera novaeangliae 26
## 6 SRR6442702 Estrecho de Magallanes Megaptera novaeangliae 21
## 7 SRR6442703 Estrecho de Magallanes Megaptera novaeangliae 37
## 8 SRR6442704 Estrecho de Magallanes Megaptera novaeangliae 32
## 9 SRR6442705 Estrecho de Magallanes Megaptera novaeangliae 28
## 10 SRR6442706 Estrecho de Magallanes Megaptera novaeangliae 30
## # ... with 77 more rows
## sample_ID geo_loc_name species observed shannon richness_0 richness_20
## <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 SRR64427... Chiloe Megapt... 63 1.28 63 63
## 2 SRR64427... Chiloe Megapt... 14 0.732 14 14
## 3 SRR64427... Chiloe Balaen... 18 1.41 18 18
## 4 SRR64427... Chiloe Balaen... 19 1.68 19 19
## 5 SRR64427... Chiloe Balaen... 36 2.31 36 36
## 6 SRR64427... Chiloe Balaen... 63 1.31 63 63
## 7 SRR64427... Chiloe Balaen... 29 1.73 29 29
## 8 SRR64427... Chiloe Balaen... 38 1.65 38 38
## 9 SRR64427... Chiloe Balaen... 81 2.05 81 81
## 10 SRR64427... Chiloe Balaen... 78 0.725 78 78 ##
# ... with 18 more rows, and 23 more variables: richness_50 <dbl>,
## # richness_80 <dbl>, diversities_inverse_simpson <dbl>,
## # diversities_gini_simpson <dbl>, diversities_shannon <dbl>,
## # diversities_fisher <dbl>, diversities_coverage <dbl>,
## # evenness_camargo <dbl>, evenness_pielou <dbl>, evenness_simpson <dbl>,
## # evenness_evar <dbl>, evenness_bulla <dbl>, dominance_dbp <dbl>,
## # dominance_dmn <dbl>, dominance_absolute <dbl>,
## # dominance_relative <dbl>, dominance_simpson <dbl>,
## # dominance_core_abundance <dbl>, dominance_gini <dbl>,
## # rarity_log_modulo_skewness <dbl>, rarity_low_abundance <dbl>,
## # rarity_noncore_abundance <dbl>, rarity_rare_abundance <dbl>
Es importante tener en cuenta que funciones provenientes de diferentes paquetes de R pueden tener el mismo nombre. Esto es altamente probable considerando que solo el repositorio CRAN contiene más de 10.000 paquetes. Por lo tanto, en ocasiones es necesario agregar el paquete fuente de la función a utilizar, así: dplyr::filter(). Con el fin de evitar errores asociados a la ejecución de una función no diferente a la deseada, pero que tiene el mismo nombre.
Para saber si el nombre función a utilizar se repite en otros paquetes, escribe en la consola el signo de interrogación seguido por el nombre de la función, así: ?filter Si el nombre se repite en diferentes paquetes, verás una lista en la pestaña Help de RStudio, sino verás la página de ayuda de la función directamente.
¿Quieres seleccionar y filtrar al mismo tiempo? Claro! Hay formas de hacer varias operaciones consecutivas en una misma instrucción, de esta manera evitamos tener que guardar objetos "intermedios" innecesariamente.
2.2 Funciones anidadas y pipes
- Vamos a anidar funciones (i.e. una función dentro de otra):
nrow(data)
## [1] 87
ncol(data)
## [1] 30
data_div <- select(filter(data, shannon > 1.5), sample_ID, geo_loc_name, species, observed, shannon)
head(data_div)
## # A tibble: 6 x 5
## sample_ID geo_loc_name species observed shannon
## <chr> <chr> < chr> <dbl> <dbl>
## 1 SRR6442697 Estrecho de Magallanes Megaptera novaeangliae 31 2.12
## 2 SRR6442699 Estrecho de Magallanes Megaptera novaeangliae 43 2.25
## 3 SRR6442703 Estrecho de Magallanes Megaptera novaeangliae 37 1.86
## 4 SRR6442708 Estrecho de Magallanes Megaptera novaeangliae 27 1.91
## 5 SRR6442709 Estrecho de Magallanes Megaptera novaeangliae 36 1.88
## 6 SRR6442712 Estrecho de Magallanes Megaptera novaeangliae 36 1.94
nrow(data_div) # 39 muestras tienen un índice de shannon mayor a 1.5## [1] 39ncol(data_div)## [1] 5
Es importante recordar que R lee la línea de comando desde dentro hacia fuera. En éste caso, primero se hizo el filtro y luego la selección.
- Usar funciones anidadas puede ser engorroso cuando quieres hacer muchas operaciones consecutivas, en cuyo caso es convenientes usar pipes. Pipes te permiten usar varias funciones consecutivas, de forma que el output de una función será en input de la siguiente. Los pipes en R lucen así: %>%.
data %>%
dplyr::filter(shannon > 1.5) %>%
dplyr::select(sample_ID, geo_loc_name, species, observed, shannon)
## # A tibble: 39 x 5
## sample_ID geo_loc_name species observed shannon
## <chr> <chr> <chr> <dbl> <dbl>
## 1 SRR6442697 Estrecho de Magallanes Megaptera novaeangli... 31 2.12
## 2 SRR6442699 Estrecho de Magallanes Megaptera novaeangli... 43 2.25
## 3 SRR6442703 Estrecho de Magallanes Megaptera novaeangli... 37 1.86
## 4 SRR6442708 Estrecho de Magallanes Megaptera novaeangli... 27 1.91
## 5 SRR6442709 Estrecho de Magallanes Megaptera novaeangli... 36 1.88
## 6 SRR6442712 Estrecho de Magallanes Megaptera novaeangli... 36 1.94
## 7 SRR6442714 Estrecho de Magallanes Megaptera novaeangli... 26 1.81
## 8 SRR6442715 Estrecho de Magallanes Megaptera novaeangli... 35 1.61
## 9 SRR6442722 Chiloe Balaenoptera musculus 19 1.68
## 10 SRR6442723 Chiloe Balaenoptera musculus 36 2.31
## # ... with 29 more rows
Como %>% pasa el objeto de su izquierda como el primer argumento de la función a su derecha, no necesitamos especificar el data frame como el primer argumento de las funciones filter() y select().
2.3 Mutate: crear nuevas columnas desde información existente en otras columnas
En algunas ocasiones necesitamos generar nueva información a partir de la existente, por ejemplo, para hacer conversiones o cálculos matemáticos.
data <- data %>%
dplyr::mutate(log10_dom_abs = log10(dominance_absolute))
# Dale un vistazo a la tabla (View(data)), la última columna es la nueva "log10_dom_abs", también notarás que, en la pestaña "Environment" de RStudio, ahora el objeto "data" tiene "31 variables".
# Si deseas tener una vista previa de como quedará tu "data" antes de guardar cualquier cambio, puedes usar pipe para agregar la función head() al final
data %>%
dplyr::mutate(log10_dom_abs = log10(dominance_absolute)) %>%
head()
## # A tibble: 6 x 31
## sample_ID geo_loc_name species observed shannon richness_0 richness_20
## <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 SRR64426... Estrecho de... Megapt... 31 2.12 31 31
## 2 SRR64426... Estrecho de... Megapt... 33 1.50 33 33
## 3 SRR64426... Estrecho de... Megapt... 43 2.25 43 43
## 4 SRR64427... Estrecho de... Megapt... 29 1.31 29 29
## 5 SRR64427... Estrecho de... Megapt... 26 1.08 26 26
## 6 SRR64427... Estrecho de... Megapt... 21 1.13 21 21
## # ... with 24 more variables: richness_50 <dbl>, richness_80 <dbl>,
## # diversities_inverse_simpson <dbl>, diversities_gini_simpson <dbl>,
## # diversities_shannon <dbl>, diversities_fisher <dbl>,
## # diversities_coverage <dbl>, evenness_camargo <dbl>,
## # evenness_pielou <dbl>, evenness_simpson <dbl>, evenness_evar <dbl>,
## # evenness_bulla <dbl>, dominance_dbp <dbl>, dominance_dmn <dbl>,
## # dominance_absolute <dbl>, dominance_relative <dbl>,
## # dominance_simpson <dbl>, dominance_core_abundance <dbl>,
## # dominance_gini <dbl>, rarity_log_modulo_skewness <dbl>,
## # rarity_low_abundance <dbl>, rarity_noncore_abundance <dbl>,
## # rarity_rare_abundance <dbl>, log10_dom_abs <dbl>
2.4 Dividir -> aplicar -> combinar
Varias operaciones de análisis de datos se pueden realizar, primero dividiendo los datos en grupos, segundo aplicando análisis a cada grupo, y tercero combinando los resultados. Hacemos esto usando las funciones group_by() y summarize() juntas. group_by() toma como argumento los nombres de la columna que contiene valores categóricos, a partir de las cuales queremos hacer algún cálculo. summarize() colapsa cada grupo en una única fila.
- Vamos a calcular el promedio y desviación estándar (mean(); sd()) del número de taxas observadas (columna "observed") por zona (columna "geo_loc_name") y especie (columna "species") de ballena:
- Si lo necesitas, también puedes agregar un filtro antes de agrupar los datos y hacer estadística de los grupos. Por ejemplo, supongamos que decidimos no considerar aquellas muestras con menos de 20 taxas (observed < 20):
- Muchas veces es útil re-organizar los datos para una más eficiente interpretación de los resultados. Por ejemplo, si queremos ordenar los resultados por número promedio de taxa observado por grupo ("mean_observed") en orden decreciente:
2.5 Contar
La función count() nos permite conocer el número de observaciones por cada variable, o combinación de ellas, en tus datos.
data %>%2.6 Remodelar tablas usando spread() y gather()
Con el objetivo de explorar las relaciones entre ciertas variables de interés en nuestros datos, podemos remodelar la tabla de datos de acuerdo a éstas variables.
Supongamos que nos interesa explorar la relación entre la diversidad del microbioma de la piel ("shannon") de las especies de ballena ("species") y su locación geográfica.
- Primero, necesitamos usar group_by() y summarize(), para agrupar nuestras variables de interés y crear una nueva columna con los valores de índice de Shannon promedio para cada grupo. Después, usamos la función spread() para transformar data de modo que: cada especie ahora sea una columna, cada locación geográfica ahora sea una fila, y los valores de Shannon estén en cada celda según corresponda.
Ahora vamos a suponer la situación contraria. Inicialmente, tenemos una tabla de datos como data_spread, en la que los nombres de las especies ("species") son columnas, pero en vez de ello, queremos que las especies sean valores de la variable "species" (columna: "species").
- Para lograrlo, necesitamos reunir los nombres de las columnas (especies) y convertirlos en un set de variables:
2.7 Exportar datos
El trabajo hecho en una sesión de R (e.g., análisis, nuevas tablas, etc.) sólo existe en la memoria de R mientras la sesión esté abierta. Necesitas exportar los nuevos datos creados para archivarlos en tu computadora. Entonces, así como existe la función read_csv() para leer archivos CSV (comma-separated values) en R, hay una función para generar archivos CSV a partir de tablas de datos en contenidas como objetos en la memoria de R.
- Por ejemplo, para exportar el data frame que creamos recién usando la función gather() y filter():
3 Visualización de datos en R
Para comenzar, primero debemos importar el o los paquetes requeridos. ggplot2 está incluido en el paquete tidyverse.
library(tidyverse)En caso de que la tabla de datos de estudio no esté en la memoria de R, puedes cargarla nuevamente.
data <- read_csv("IR_table1.csv")ggplot2 es un paquete para graficar, que facilita crear gráficas complejas a partir de datos en un data frame. Incluye varias funciones para especificar que variables graficar, como éstas son expuestas, y varias otras características visuales. ggplot2 funciona mejor con datos extensos, i.e., una columna por cada dimensión, y una fila por cada observación. Tener datos bien estructurados te ayudará a crear figuras más eficientemente.
3.1 Graficar con ggplot2
Las gráficas de ggplot son construidas paso a paso, agregando nuevos elementos cada vez. Para graficar con ggplot, vamos a usar una línea de comando templado que es útil para diferentes tipos de gráficos:
ggplot(data = , mapping = aes()) + ()
Usamos la función ggplot() y el argumento data para indicar a partir de qué datos se debe crear la gráfica. Luego, la función aes() (aesthetic) para seleccionar las variables a graficar y como presentarlas, e.g. ejes x e y o características como tamaño, forma, color, etc.
ggplot2 ofrece "geoms" para indicar la representación gráfica de los datos (puntos, líneas, barras), algunos de ellos son:
- geom_point() para gráficos de dispersión.
- geom_boxplot() para gráficos de caja (boxplots).
geom_line() para líneas de tendencia, series de tiempo, etc.
Usa + para agregar un geom a línea de comando de ggplot.
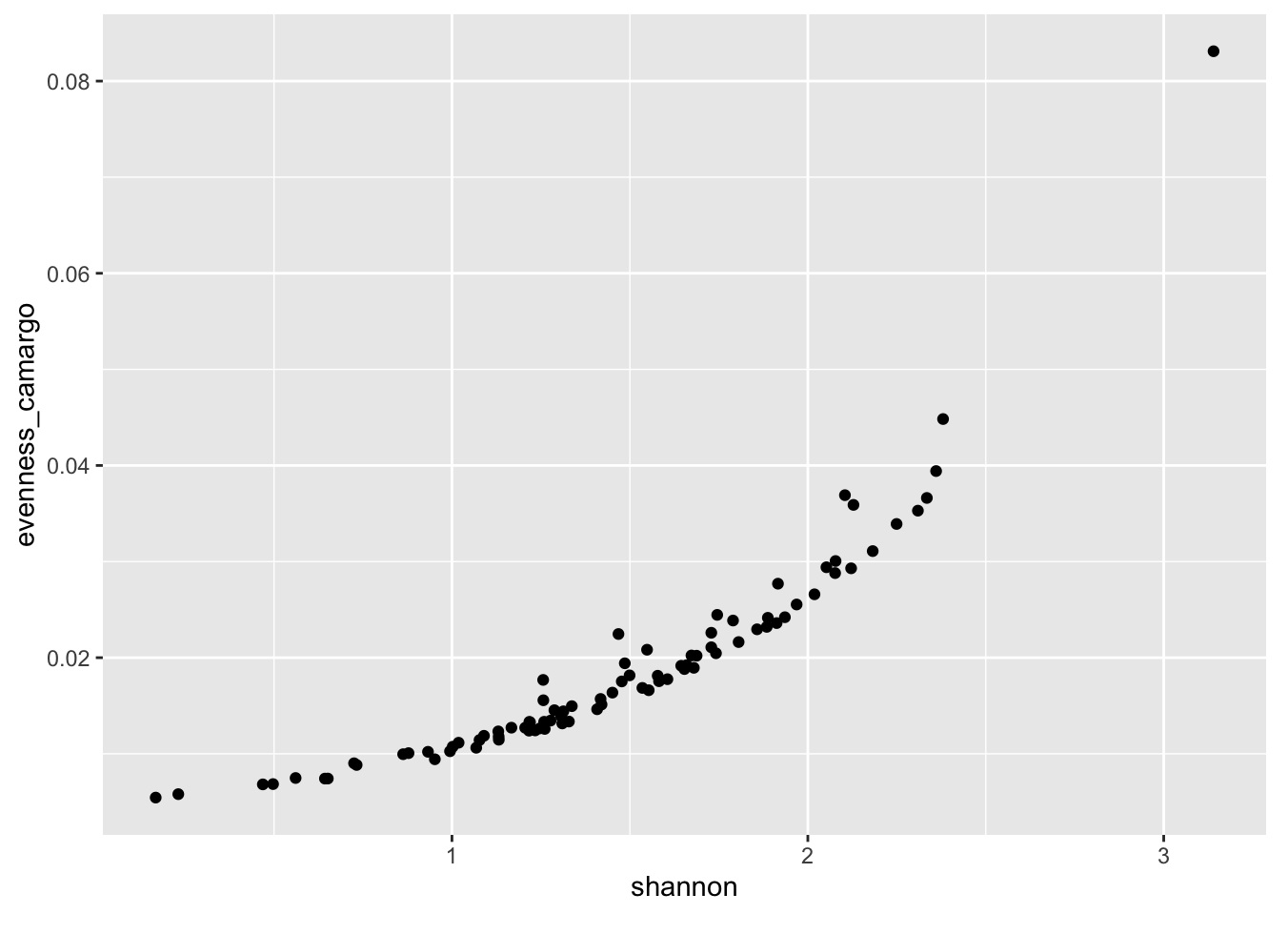
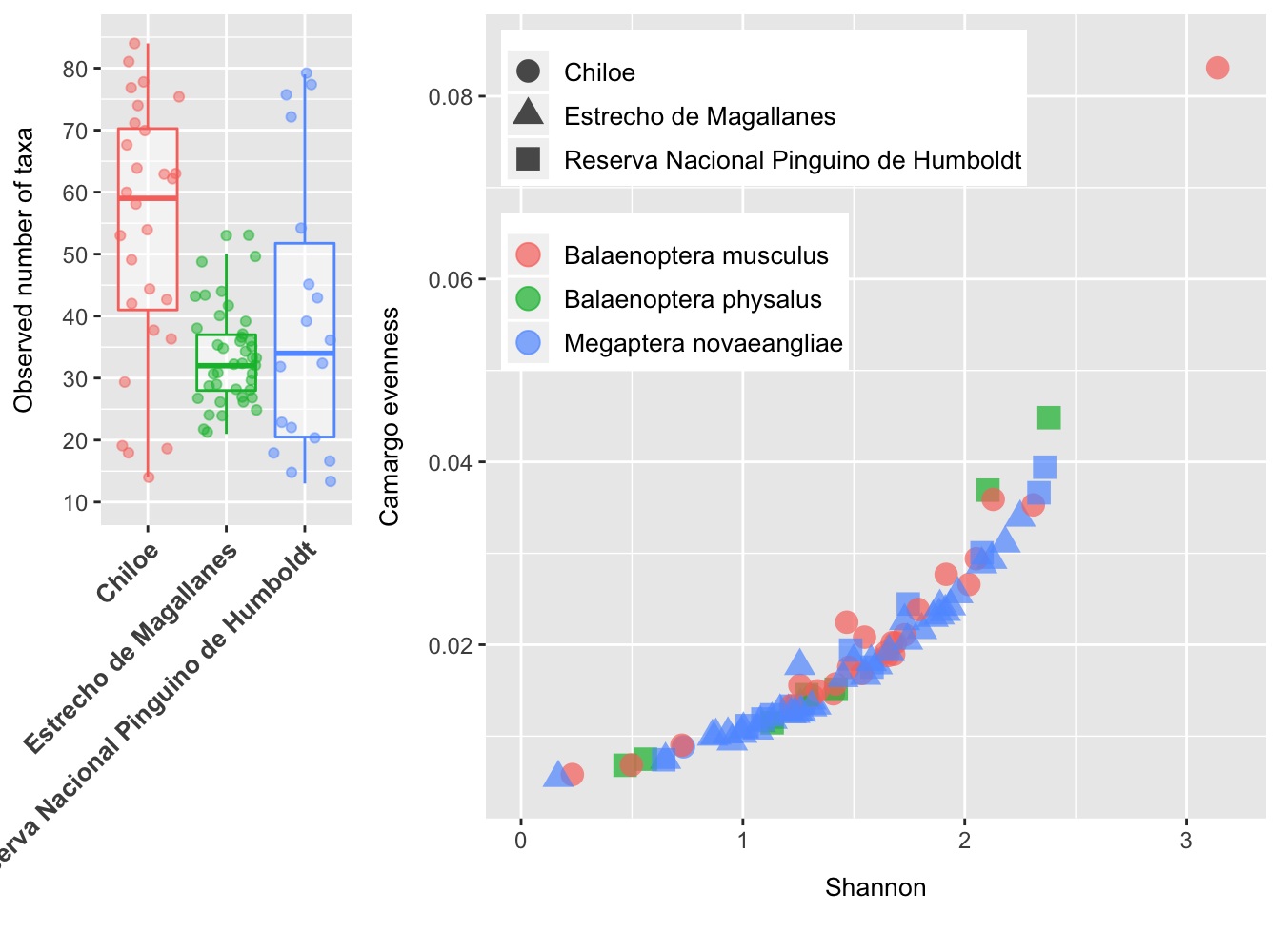
ggplot(data = data, aes(x = shannon, y = evenness_camargo)) + geom_point()
Título
Aquí comienza tu texto. Puedes hacer clic en este punto y empezar a escribir. Quae ab illo inventore veritatis et quasi architecto beatae vitae dicta sunt explicabo nemo enim ipsam voluptatem quia voluptas sit aspernatur aut odit aut fugit sed quia.

Seguidamente, empezamos a modificar el gráfico para extraer más información de él y hacerlo más explíito o auto-explicativo. Por ejemplo, podemos agregar transparencia (alpha) para evidenciar la sobre-posición de los puntos (overplotting). También podemos colorear los puntos (color).
ggplot(data = data, aes(x = observed, y = shannon)) + geom_point(alpha = 0.4, color = "blue")
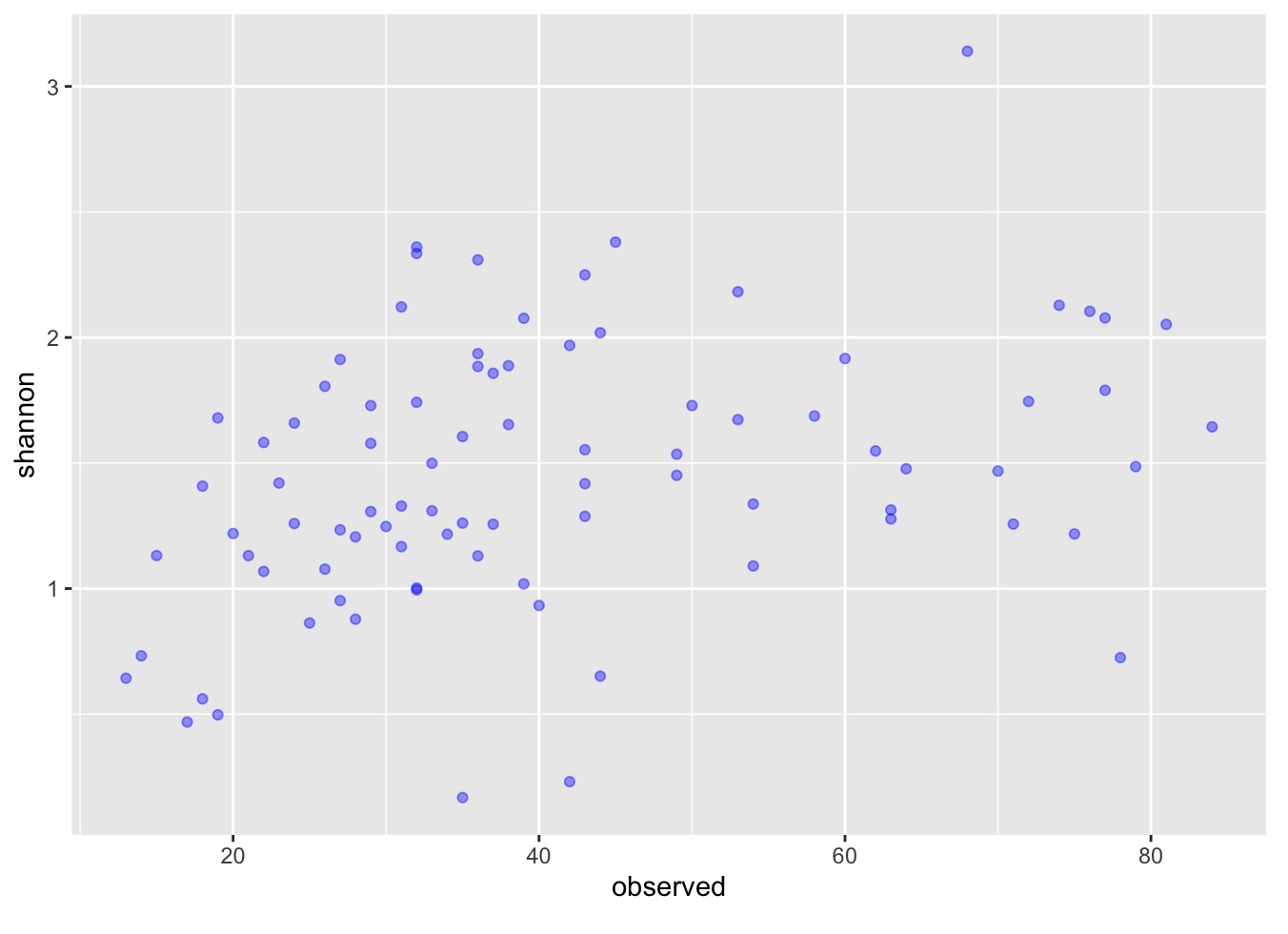
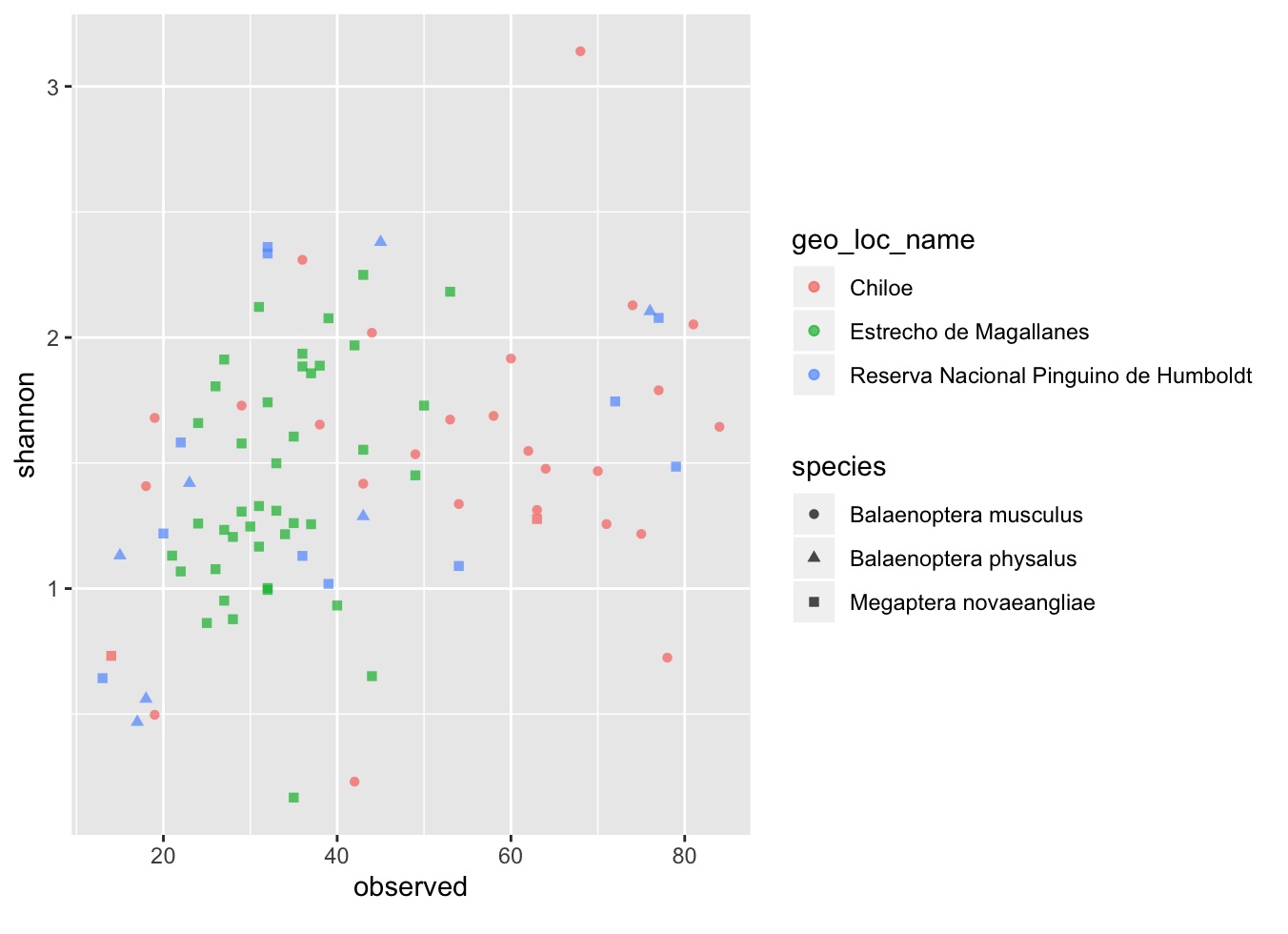
Además, es posible "anotar" tus datos de acuerdo a cierta(s) variable(s) categórica(s), es decir, colorearlos o asignarles formas según ciertas características que tu consideres influyentes en tus datos. Por ejemplo, vamos a anotar nuestra gráfica por especie ("species") y por locación geográfica ("geo_loc_name").
ggplot(data = data, aes(x = observed, y = shannon)) + geom_point(alpha = 0.7, aes(color = geo_loc_name, shape = species))
3.3 Boxplot
Boxplots o gráficos de caja, son útiles para visualizar la distribución de los datos de acuerdo a una variable o condición de interés.
- Por ejemplo, para visualizar la distribución de número de taxas observadas en el microbioma de la piel de las ballenas muestreadas en Chiloé, Estrecho Magallanes y Parque Nacional Pingüino de Humboldt:
ggplot(data = data, aes(x = geo_loc_name, y = observed)) +
geom_boxplot()
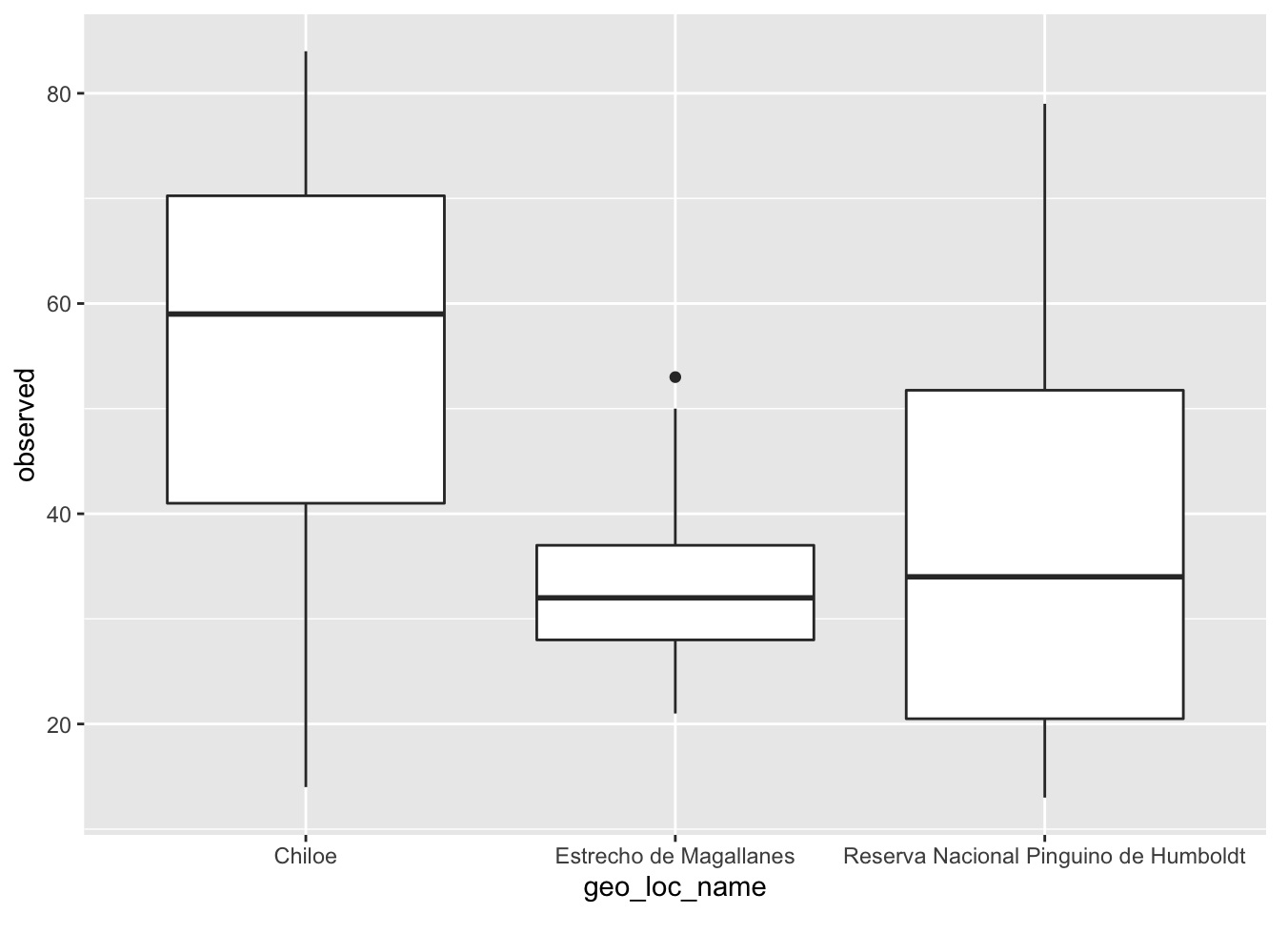
- Sin embargo, ¿podría éste resultado estar sesgado por el tamaño muestreal de cada locación geográfica?. Veamos cuántas muestras tenemos por locación geográfica:
data %>% dplyr::count(geo_loc_name, sort = TRUE)
- Agregar puntos al boxplot nos da una mejor idea del número de muestras y su distribución:
ggplot(data = data, aes(x = geo_loc_name, y = observed)) +
geom_boxplot(alpha = 0.5) +
geom_jitter(alpha = 0.5, color = "tomato")
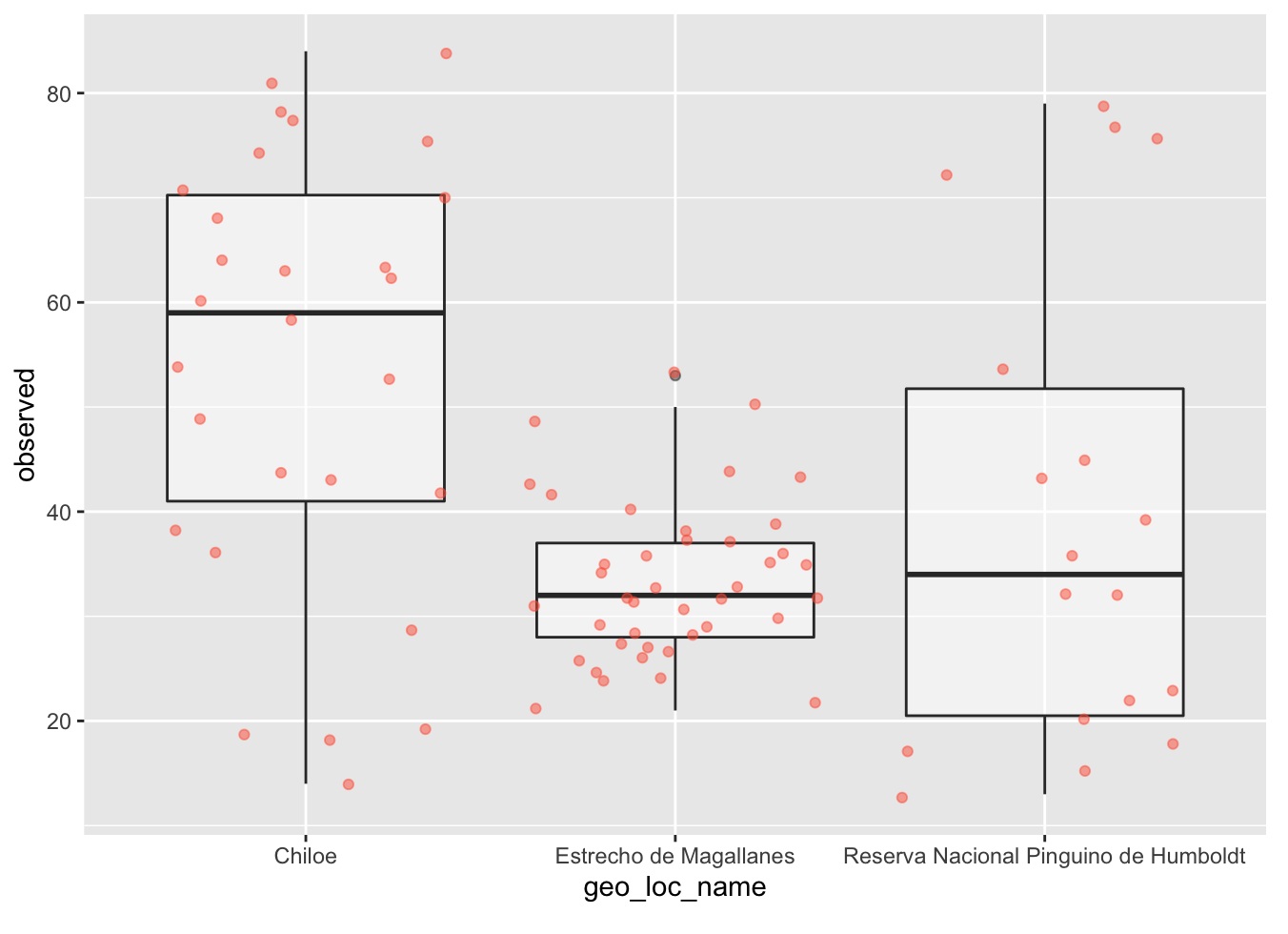
¿Notas como las cajas (boxplot layer) están detrás de los puntos (jitter layer)? ¿Qué necesitas cambiar en el código de ggplot para que las cajas aparezcan en frente de los puntos? (Pista: el orden sí importa)
3.4 Graficar datos con series de tiempo
Cuando tienes datos tomados en una serie de tiempo, una buena manera de visualizarlos es a través de un gráfico de líneas. En el siguiente ejemplo vamos a utilizar una tabla de datos correspondiente a un sub-muestreo de un experimento RNAseq, que contiene las read counts de 8 transcritos después de 0, 3, 6, 12 y 24 horas de exposición a un estímulo X.
Descarga la tabla de datos VD_table2.tbl
Primero vamos a cargar dicha tabla a la memoria de R:
tr_counts <- read_table2(file = "~/Dropbox/CastroLab_database/workshops_data/VD_table2.tbl", col_names = TRUE)
## Parsed with column specification:
## cols(
## transcripts = col_character(),
## `0hrs` = col_double(),
## `3hrs` = col_double(),
## `6hrs` = col_double(),
## `12hrs` = col_double(),
## `24hrs` = col_double() ## )
# Usamos la función read_table2() para leer el archivo `data/VD_table2.tbl` y asignarlo al nuevo objeto `tr_counts`.
# El argumento col_names = TRUE es para indicar la presencia de títulos o nombres para cada columna.
tr_counts # dale un vistazo a la nueva tabla de datos
## # A tibble: 8 x 6
## transcripts ` 0hrs` `3hrs` `6hrs` `12hrs` `24hrs`
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 Transcript_1 2.67 24.5 59.2 9.00 14.2
## 2 Transcript_2 0 87.0 5.71 7.61 0
## 3 Transcript_3 1.84 54.9 131. 63.5 15.6
## 4 Transcript_4 0.256 16.6 27.1 1.33 18.5
## 5 Transcript_5 0 0 0 0 0
## 6 Transcript_6 0 6.31 0 0 0
## 7 Transcript_7 0 6.64 0 0 0
## 8 Transcript_8 0 767. 92.7 141. 0
Generalmente, a través de la metodología de RNAseq, podemos obtener una tabla como tr_counts, que informa de las read counts de cada transcrito por tiempo muestral. Sin embargo, para crear un gráfico lineal, usando ggplot() + geom_line(), en el que el eje X represente una serie de tiempo, es necesario que los tiempos muestreales sean una variable (no nombres de columnas, como en tr_counts).
- Entonces, recordamos lo aprendido anteriormente y utilizamos la función gather() para remodelar tr_counts, reuniendo los nombres de las columnas (tiempos) y transformándolos en un set de variables:
tr_plot <- tr_counts %>% gather(key = time, value = read_counts, -transcripts) # View(tr_plot)
- Ahora podemos graficar las read counts de cada transcrito a través de los 5 puntos muestreales, usando ggplot() + geom_line():
ggplot(data = tr_plot, aes(x = time, y = read_counts, group = transcripts)) +
geom_line()
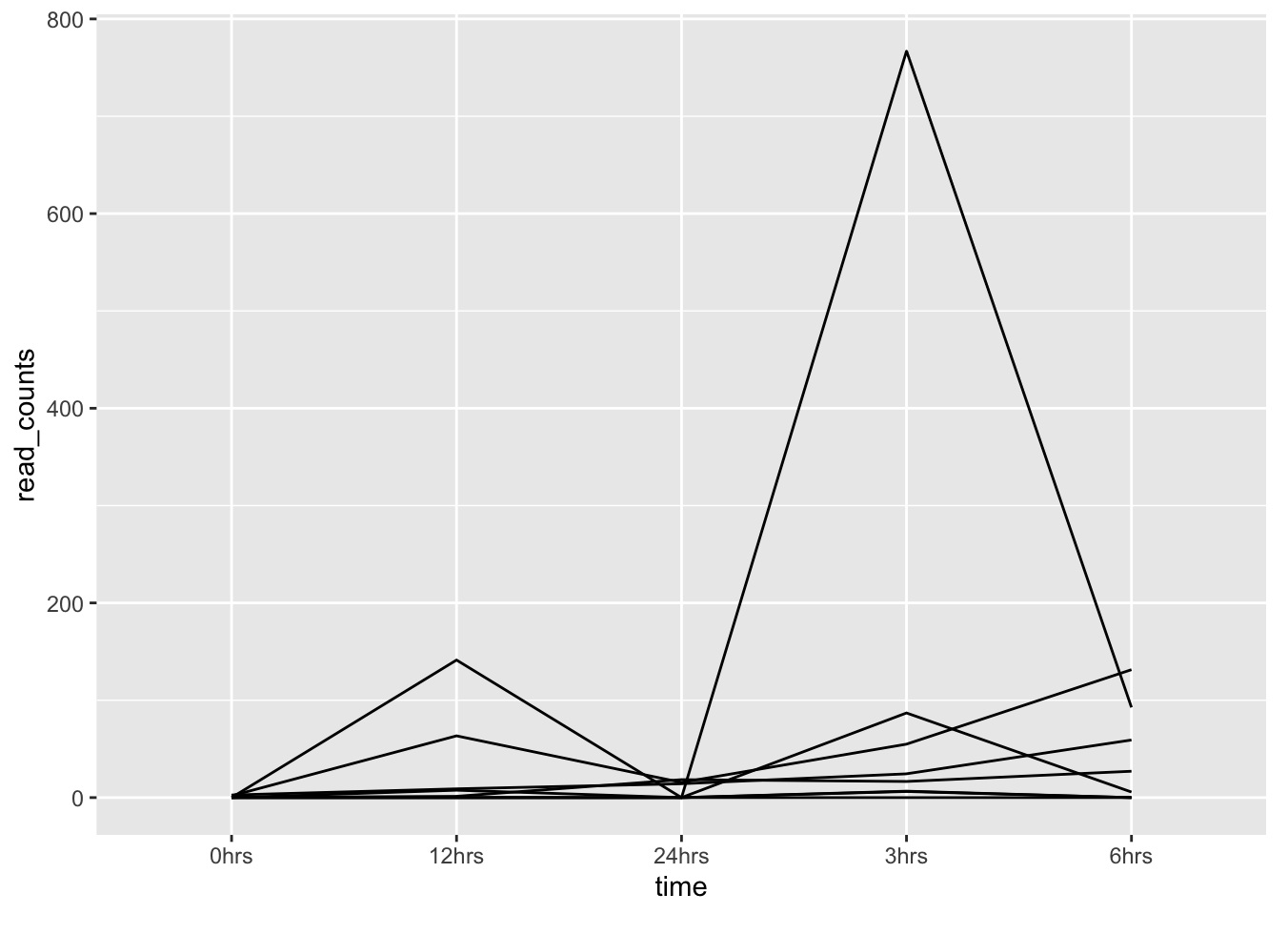
- ¿Aún no? Podemos anotar las líneas para identificar cada transcrito:
ggplot(data = tr_plot, aes(x = time, y = read_counts, group = transcripts)) +
geom_line(aes(color = transcripts))
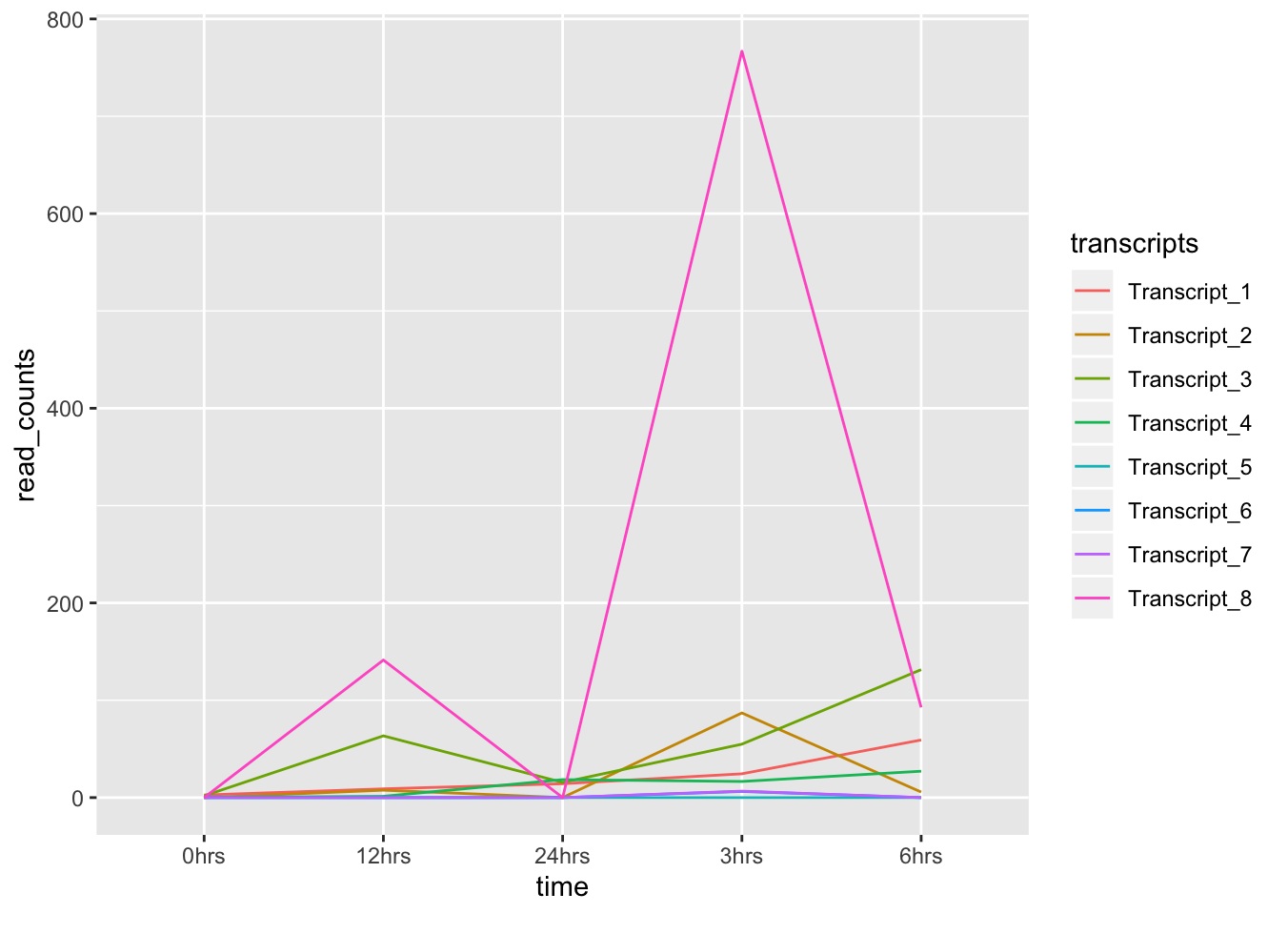
Como ya habrás notado, tanto en la gráfica como en la tabla, el rango de los valores de read counts es bastante amplio...
min(tr_plot$read_counts) # 'objeto$columna'
## [1] 0
max(tr_plot$read_counts)
## [1] 766.8096
Al graficar, en muchos cachos es conveniente considerar la escala de las observaciones, con el fin de obtener una mejor visualización de la distribución de los datos.
- Vamos a convertir los valores de read counts al logarítmo base 10 de las read counts, de modo de reducir el rango de distribución de los valores. Para ello, modificamos la escala del eje Y usando scale_y_log10():
ggplot(data = tr_plot, aes(x = time, y = read_counts, group = transcripts)) + geom_line(aes(color = transcripts)) + scale_y_log10()
## Warning: Transformation introduced infinite values in continuous y-axis

¡Warning message! Obtenemos un gráfico, pero algo no está bien...
¿A qué crees que se debe el mensaje de advertencia? ¿Qué otro aspecto del gráfico crees tú es necesario modificar?
- Solución:
ggplot(data = tr_plot, aes(x = time, y = log10(read_counts+1), group = transcripts)) +
geom_line(aes(color = transcripts))
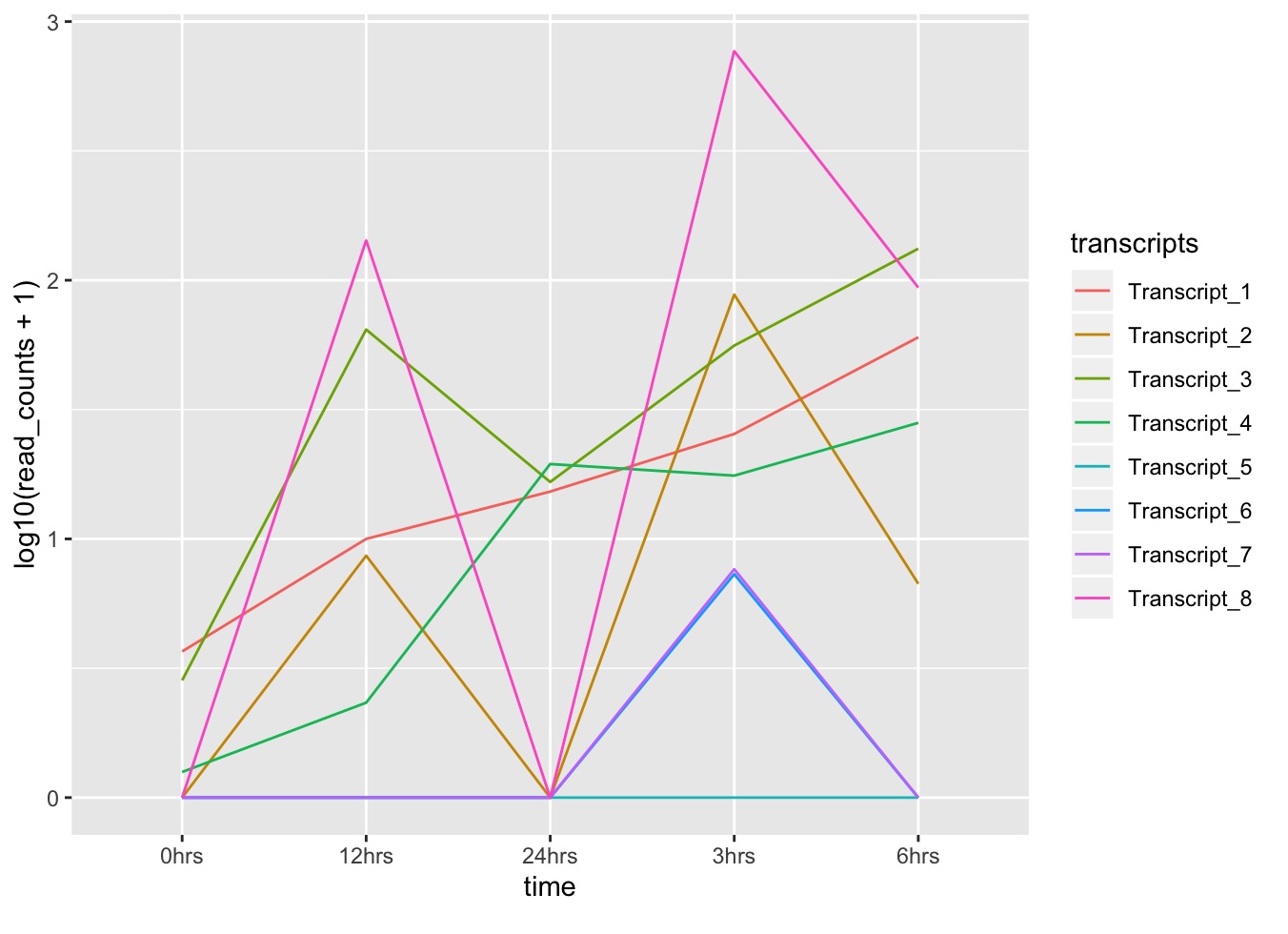
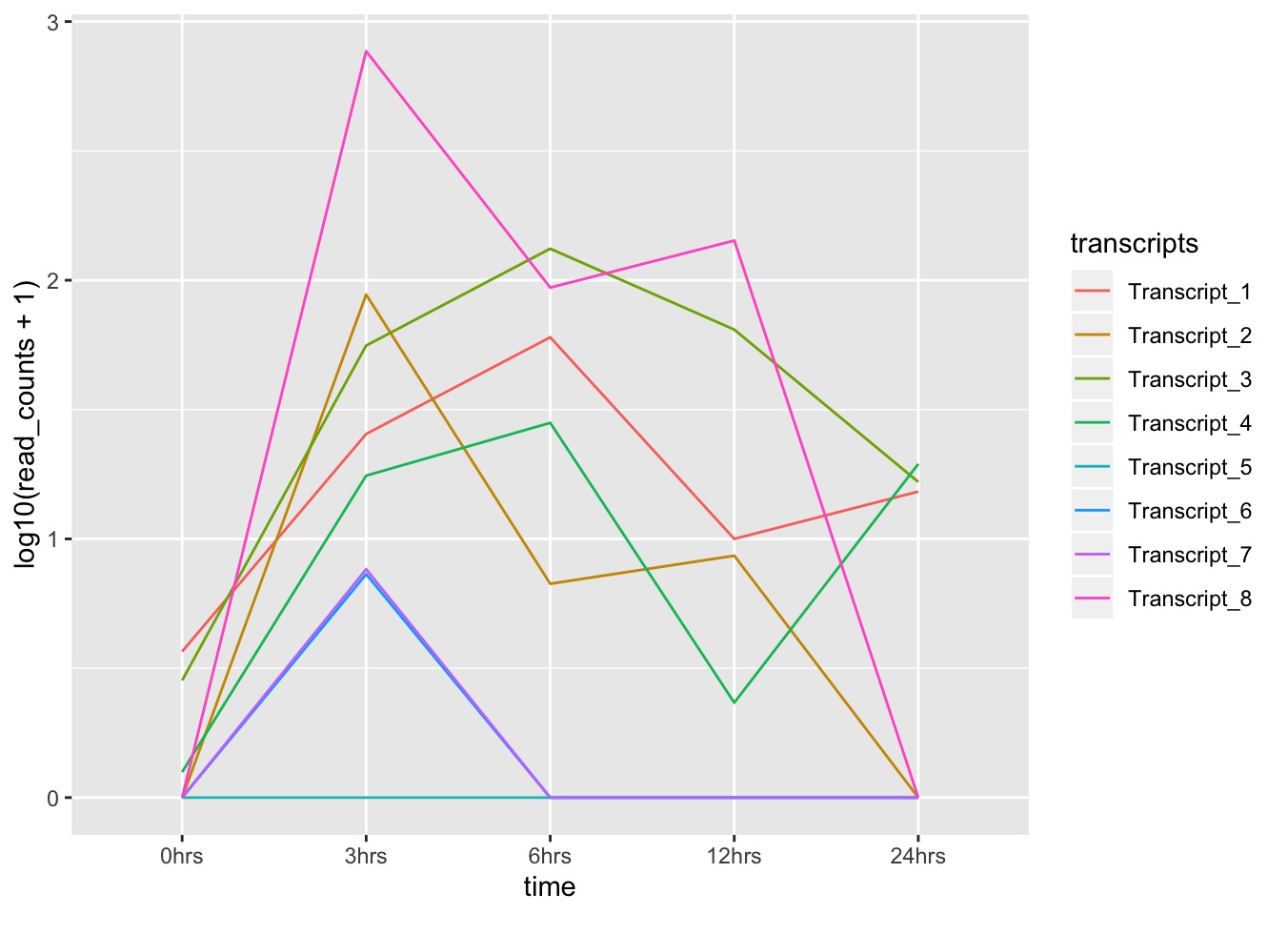
¡Truco! Transforma la columna "time" en un factor ordenado, para que ggplot respete el orden deseado de tu serie de tiempo:
tr_plot$time <- factor(tr_plot$time, levels=unique(tr_plot$time))
ggplot(data = tr_plot, aes(x = time, y = log10(read_counts+1), group = transcripts)) +
geom_line(aes(color = transcripts))
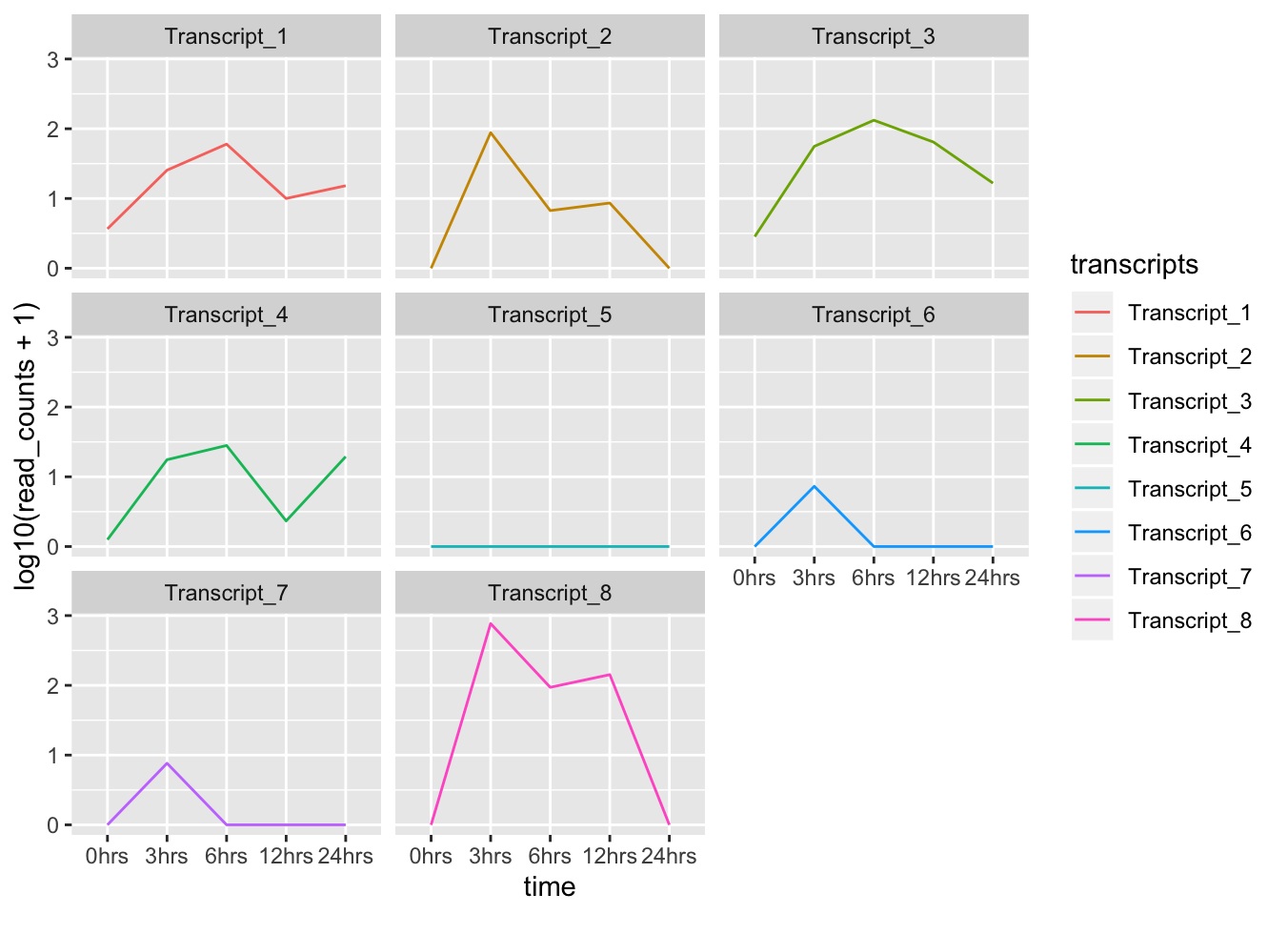
3.5 Faceting
Faceting es una propiedad de ggplot que permite dividir un gráfico en múltiples gráficos basado en un factor incluído en el set de datos.
- Continuando con el gráfico que hemos estado trabajando, usamos facet_wrap() para hacer un gráfico por cada transcrito:
ggplot(data = tr_plot, aes(x = time, y = log10(read_counts+1), group = transcripts)) +
geom_line(aes(color = transcripts)) +
facet_wrap(~transcripts)
3.6 ggplot2 themes
Themes permite configurar la apariencia o características estéticas de los gráficos creados con ggplot. Puedes revisar la lista completa de themes disponibles aquí.
- Por ejemplo, gráficos con fondo blanco lucen mejor cuando son impresos. Podemos hacer que el fondo de las gráficas sea blanco usando la función theme_bw(), también podemos remover la cuadrícula:
ggplot(data = tr_plot, aes(x = time, y = log10(read_counts+1), group = transcripts)) +
geom_line(aes(color = transcripts)) +
facet_wrap(~ transcripts) +
theme_bw() +
theme(panel.grid = element_blank())
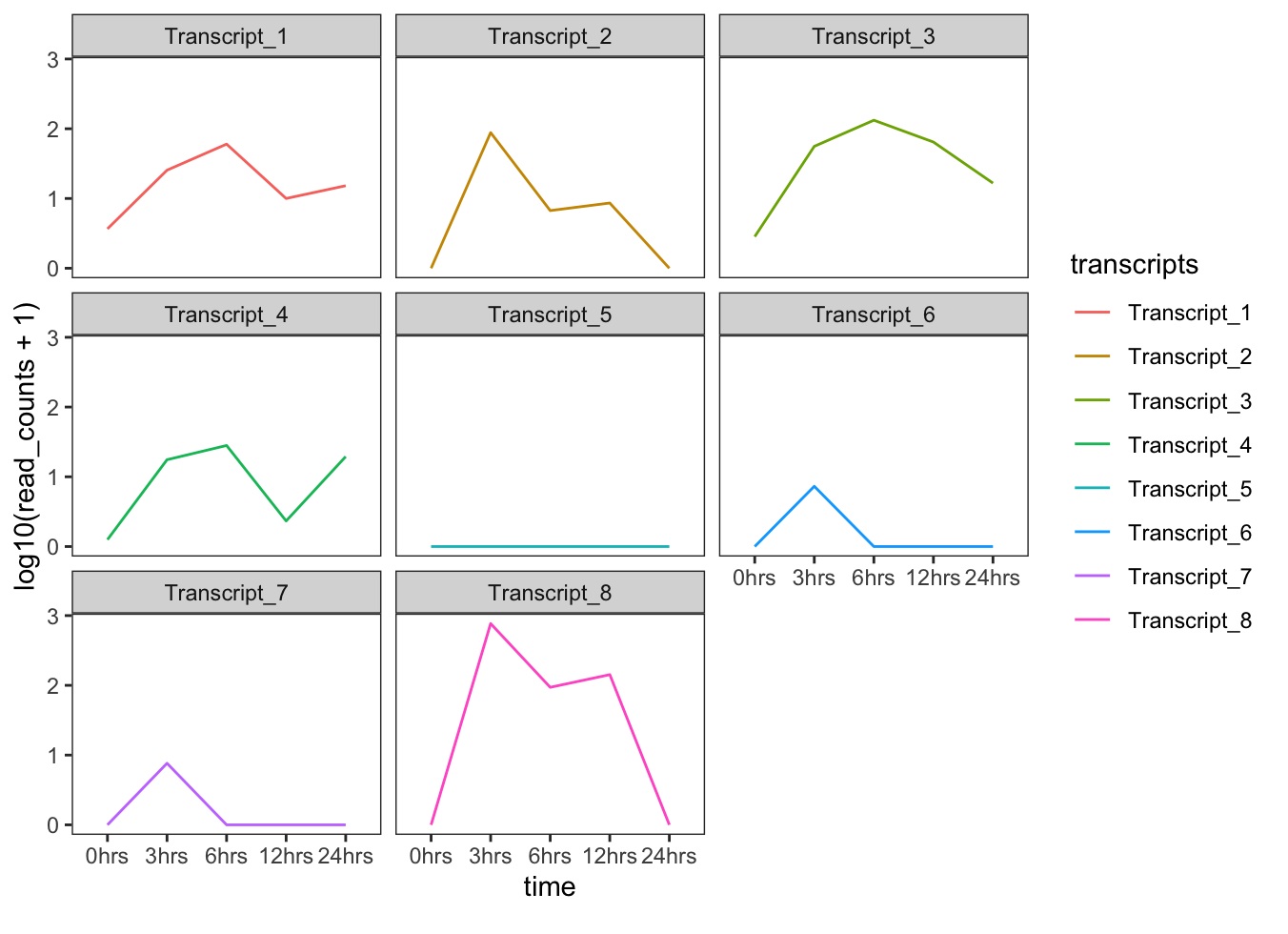
Mientras que, la función facet_wrap() organiza los gráficos en un número arbitrario de filas y columnas, también existe la función facet_grid que permite especificar como organizar los gráficos usando la siguiente nomenclatura: filas ~ columnas, también podemos usar . para indicar sólo una fila o columna (e.g., todas las gráficas en una columna: facet_grid(transcripts ~ .)).
3.7 Personalización
ggplot2 cuenta con varias funciones para personalizar gráficas, dale un vistazo al Cheat Sheet "Data Visualization" para pensar en formas de mejorar tus gráficos, también puedes inspirarte buscando ejemplos en internet, como aquí.
- Vamos a revisar un ejemplo de como personalizar el boxplot que estuvimos trabajando anteriormente:
ggplot(data = data, aes(x = geo_loc_name, y = observed, color = geo_loc_name)) +
geom_boxplot(alpha = 0.5) +
geom_jitter(alpha = 0.5) +
coord_cartesian(ylim = c(10, 85)) +
scale_y_continuous(breaks = c(10,20,30,40,50,60,70,80)) +
theme(axis.text.x = element_text(face = "bold", angle = 45, hjust = 1, size = 10),
axis.title.x = element_blank(),
axis.text.y = element_text(size = 9),
axis.title.y = element_text(size = 10, margin = margin(t = 0, r = 10, b = 0, l = 0)),
legend.position = "none") +
labs(y = "Observed number of taxa")

3.8 Organizar y exportar gráficos
Anteriormente usamos faceting para separar un gráfico en múltiples gráficos. Sin embargo, también podemos crear una figura con múltiples gráficos diferentes (i.e., diferentes variables, set de datos, tipo de gráfica). El paquete gridExtra nos permite combinar gráficos separados en una única figura, usando la función grid.arrange().
p1 <- ggplot(data = data, aes(x = geo_loc_name, y = observed, color = geo_loc_name)) +
geom_boxplot(alpha = 0.5) +
geom_jitter(alpha = 0.5) +
coord_cartesian(ylim = c(10, 85)) +
scale_y_continuous(breaks = c(10,20,30,40,50,60,70,80)) +
theme(axis.text.x = element_text(face = "bold", angle = 45, hjust = 1, size = 10),
axis.title.x = element_blank(),
axis.text.y = element_text(size = 9),
axis.title.y = element_text(size = 10, margin = margin(t = 0, r = 10, b = 0, l = 0)),
legend.position = "none") +
labs(y = "Observed number of taxa")
p2 <- ggplot(data = data, aes(x = shannon, y = evenness_camargo, color = species, shape = geo_loc_name)) +
geom_point(alpha = 0.7, size = 4) +
coord_cartesian(xlim = c(0.0,3.2), ylim = c(0.005,0.085)) +
theme(axis.text.x = element_text(size = 9),
axis.title.x = element_text(size = 10, margin = margin(t = 10, r = 0, b = 0, l = 0)),
axis.text.y = element_text(size = 9),
axis.title.y = element_text(size = 10, margin = margin(t = 0, r = 10, b = 0, l = 0)),
legend.text = element_text(size = 10),
legend.title = element_blank(),
legend.position = c(.02, .98),
legend.justification = c("left", "top"),
legend.box.just = "left",
legend.margin = margin(2, 2, 2, 2)) +
labs(x = "Shannon", y = "Camargo evenness")
grid.arrange(p1, p2, ncol = 2, widths = c(1,2.5))
Después de crear tu gráfico o figura, puedes guardarlo en un archivo de formato de preferencia (e.g., png, pdf):
En RStudio, dirígete a la pestaña Plots y haz clic en Export para guardar el gráfico que estés visualizando.
También puedes usar la función ggsave() para, no solo guardar tus gráficos, sino que también modificar la dimensión y resolución por medio de los argumentos width, height y dpi.
# Asigna el gráfico a un 'objeto'
plot <- grid.arrange(p1, p2, ncol = 2, widths = c(1,2.5), heights = c(3,1))
# Exporta la nueva figura en fig_output/
ggsave("example_plot.pdf", plot, width = 14, height = 8, device = "pdf")
Análisis de secuencias de 16S con DADA2
1 Introducción al análisis por 16S rRNA con DADA2 y mothur
Lo primero que tenemos que hacer es instalar y cargar los paquetes que vamos a usar en esta sección. Algunos están alojados en el repositorio CRAN https://cran.rediris.es/ , otros en el repositorio Bioconductor, https://www.bioconductor.org/ y otros en el repositorio GitHub https://github.com/
1.1 Configurar sesión de R
Para trabajar en R, hay tres primeros pasos que debemos seguir: (1) cargar a los paquetes necesarios a la sesión actual, (2) configurar el directorio de trabajo, (3) importar datos de entrada a la sesión actual.
Los siguientes 3 scripts te mostrarán una manera eficiente de instalar y cargar la lista de paquetes según su repositorio de origen, ya que cada repositorio tiene su propia función para instlar sus paquetes.
- Primero, enlistar los paquetes necesarios en diferentes vectores dependiendo de su repositorio de origen.
# Definir paquetes
## Repositorio CRAN
cran_packages <- c("bookdown", "knitr", "tidyverse", "plyr", "grid", "gridExtra", "kableExtra", "xtable", "ggpubr")
## Repositorio Bioconductor
bioc_packages <- c("phyloseq", "dada2", "DECIPHER", "phangorn", "ggpubr", "BiocManager","DESeq2", "microbiome", "philr")
## Repositorio GitHub
git_source <- c("twbattaglia/btools", "gmteunisse/Fantaxtic", "MadsAlbertsen/ampvis2", "opisthokonta/tsnemicrobiota")
# fuente/nombre del paquete
git_packages <- c("btools", "fantaxtic", "ampvis2", "tsnemicrobiota") # nombre del paquete
- Segundo, instalar los paquetes definidos arriba usando la función correspondiente a cada repositorio.
# Instalar paquetes CRAN
.inst <- cran_packages %in% installed.packages()
if(any(!.inst)) { install.packages(cran_packages[!.inst]) }
# Intalar paquetes BioConductor
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
.inst <- bioc_packages %in% installed.packages()
if(any(!.inst)) {
BiocManager::install(bioc_packages[!.inst]) }
# Instalar paquetes GitHub
.inst <- git_source %in% installed.packages()
if(any(!.inst)) {
devtools::install_github(git_source[!.inst]) }
- Tercero, cargar los paquetes requeridos a la sesión actual de R.
# Cargar paquetes sapply(c(cran_packages, bioc_packages, git_packages), require, character.only = TRUE)
El paso de instalación de paquetes es necesario solamente una vez. Excepto si se quiere actualizar la versión del paquete, o bien, R ha sido desinstalado e instalado nuevamente o actualizado su versión.
Si ya tienes los paquetes instalados en tu computadora, sólo necesitas enlistar (1er script) y cargar (3er script) los paquetes. También, puedes cargarlos a la sesión actual de R usando la función library(), así:
# Cargar paquetes
library(tidyverse)
library(plyr)
library(grid)
library(gridExtra)
library(kableExtr)
library(xtable)
library(ggpubr)
library(phyloseq)
library(dada2)
library(DECIPHER)
library(phangorn)
library(ggpubr)
library(BiocManager)
library(DESeq2)
library(microbiome)
library(philr)
library(btools)
library(fantaxtic)
library(ampvis2)
library(tsnemicrobiota)
1.2 DADA2
Este tutorial se basa fuertemente en el trabajo de otros investigadores publicado en Callahan BJ, Sankaran K, Fukuyama JA et al. https://www.ncbi.nlm.nih.gov/pubmed/27508062
En esta sección vamos a explorar un set de datos de 98 muestras de piel de ballenas, jorobadas, azules y otras. Las muestras fueron obtenidas al amplificar y secuenciar un fragmento de la región variable 4 (V4) del gen 16S rRNA gene. Las secuencias están disponibles en este link
https://www.dropbox.com/sh/vkc1p96ney0x6kh/AADjo1ayo3ic5OhzNE2WRac1a?dl=0
y también en la base de datos de NCBI SRA bajo el número de acceso
https://www.ncbi.nlm.nih.gov/bioproject/PRJNA428495
Entonces, lo que vamos a hacer es correr nuestros datos según la pipeline DADA2. DADA2 ofrece ventajas con respecto a la estrategia de formar clusters (OTUs) en varios aspectos que incluyen mayor resolución, nombres consistentes entre diferentes análisis, mejor estimación de abundancias relativas, etc. Los siguientes artículos ofrecen una discusión más detallada al respecto ( artículo y artículo).
Lo primero que vamos a hacer es descargar las reads que están contenidas en la carpeta whale_pe/, descárgala https://www.dropbox.com/s/es3ehm61z0hlmb6/whale_pe.zip?dl=0
Ahora, vamos a configurar el directorio donde están las reads:
set.seed(100)
Asegúrate de modificar la ruta: ~/Dropbox/CastroLab_database/workshops_data/whale_pe/ a la ruta de directorios según tu computadora.
- En DADA2 las reads se trabajan inicialmente por separado, es decir, la copia forward o R1 separada de la copia reverse o R2. Por esto, nos tenemos que asegurar que ambos archivos estén ordenados. Luego, manipulamos el nombre de los archivos para generar automáticamente el nombre de las muestras en nuestro análisis:
fnFs <- sort(list.files(miseq_path, pattern="_1.fastq")) fnRs <- sort(list.files(miseq_path, pattern="_2.fastq"))
- El paquete DADA2 tiene muchas funciones útiles para el preprocesamiento de los datos. Acá lo que hacemos es visualizar el perfil de calidad de las muestras para cada par de 5' a 3':
plotQualityProfile(fnFs[1:2])
plotQualityProfile(fnRs[1:2])
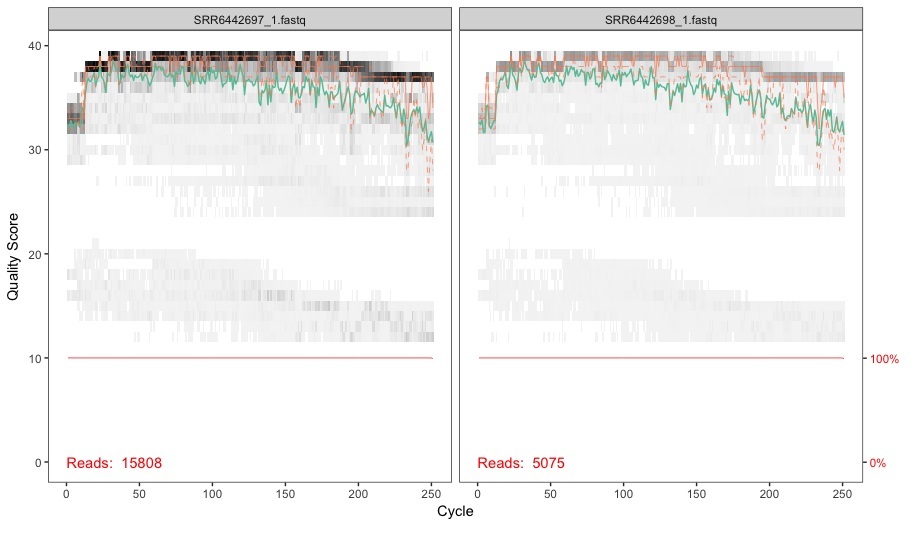
- Ahora vamos a proceder con el filtrado y corte de las reads de acuerdo a lo observado en los gráficos de calidad. Primero creamos un directorio donde vamos a poner las reads una vez realizado el control de calidad, y luego realizamos el dicho control. En específico usamos los argumentos trunLen =, maxN=, maxEE= y rm.phix= para indicar el corte promedio de cada read, el número máximo de bases indeterminadas, el número máximo de errores, y si es que queremos remover secuencias pertenecientes al control interno de Illumina.
# Creamos un directorio para poner las reads "limpias" filt_path <- file.path(miseq_path, "filtered") if(!file_test("-d", filt_path)) dir.create(filt_path) filtFs <- file.path(filt_path, paste0(sampleNames, "_F_filt.fastq.gz")) filtRs <- file.path(filt_path, paste0(sampleNames, "_R_filt.fastq.gz")) # Y finalmente procedemos con el control de calidad out <- filterAndTrim(fnFs, filtFs, fnRs, filtRs, truncLen=c(250,200), maxN=0, maxEE=c(2,2), truncQ=2, rm.phix=TRUE, compress=TRUE, multithread=TRUE) # Si usan Windows, configuren multithread=FALSE
- DADA2 genera un modelo probabilístico de errores con el cual puede filtrar reads erróneas y así usar las restantes directamente para la etapa de clasificación taxonómica. Esta parte del método es la que nos permite tener una mayor resolución en comparación a los análisis basados en OTUs. Como las muestras probablemente tienen reads idénticas, no es eficiente usar cada una de ellas para los pasos río abajo. Por esto, DADA2 recomienda de-replicar las muestras para así disminuir la redundancia y avanzar eficientemente.
derepFs <- derepFastq(filtFs, verbose=TRUE) derepRs <- derepFastq(filtRs, verbose=TRUE) # Acá simplemente agregamos los nombres de las muestras al objeto de-replicado names(derepFs) <- sampleNames names(derepRs) <- sampleNames
- Una vez con las muestras de-replicadas procedemos a generar un modelo de errores. Para mayor detalle sobre este crucial paso revisen el artículo original aquí.
errF <- learnErrors(filtFs, multithread=TRUE)
errR <- learnErrors(filtRs, multithread=TRUE)
# Graficamos los errores para cada par
plotErrors(errF)
plotErrors(errR)

Cada panel en el gráfico nos indica la frecuencia de error de nuecleótido a nucleótido para todas las combinaciones. Naturalmente, las bases con mayor puntaje de calidad exhiben una menor frecuencia de error.
- Con esta información entonces procedemos al paso más importante de la pipeline, i.e., la inferencia de las Amplicon Sequence Variants (ASVs).
dadaFs <- dada(derepFs, err=errF, multithread=TRUE, pool = TRUE) dadaRs <- dada(derepRs, err=errR, multithread=TRUE, pool = TRUE)
- Ahora nos queda hacer un poco de aseo: primero unimos las reads R1 y R2, luego generamos una tabla de secuencias que vamos a utilizar más tarde, y finalmente removemos secuencias artefactuales producto de la amplificación por PCR.
# Unimos las reads mergers <- mergePairs(dadaFs, derepFs, dadaRs, derepRs) # Generamos una tabla de secuencias seqtabAll <- makeSequenceTable(mergers[!grepl("Mock", names(mergers))]) table(nchar(getSequences(seqtabAll))) # Removemos las secuencias quiméricas seqtabNoC <- removeBimeraDenovo(seqtabAll)
La tabla de secuencias sin las quimeras, es la tabla que usamos para realizar la clasificación taxonómica. En principio, podríamos usar cualquiera de las tres bases de datos más populares para clasificación de secuencias del 16S rRNA, i.e., GreenGenes, RDP o SILVA.
En nuestro práctico vamos a utilizar SILVA, en particular, la versión 132.
Antes de continuar, descarga los archivos de la base de datos SILVA que vamos a utilizar: silva_nr_v132_train_set.fa AQUÍ y silva_species_assignment_v132.fa AQUÍ.
fastaRef <- "~/Dropbox/CastroLab_database/workshops_data/silva_nr_v132_train_set.fa" taxTab <- assignTaxonomy(seqtabNoC, refFasta = fastaRef, multithread=TRUE) # En el caso de querer agregar el rango taxonómico de especies, simplemente usamos una base de datos extra, la cual contiene esta información. taxTabExtra <- addSpecies(taxTab, "~/Dropbox/CastroLab_database/workshops_data/silva_species_assignment_v132.fa", verbose=TRUE) unname(head(taxTab)) -> tabla colnames(tabla) <- c("Kingdom", "Phylum", "Order", "Class", "Family", "Genus")
Ya que nuestro perfil taxonómico se construye con secuencias homólogas del 16S rRNA gene, podemos usar estas secuencias para inferir un árbol filogenético. Existen muchos paquetes de r que pueden hacer esto y aquí escogemos phangorn para hacer una inferencia basada en Maximum Likelihood.
- Primero alineamos las secuencias:
seqs <- getSequences(seqtabNoC) # Este paso propaga los nombres de las secuencias al árbol names(seqs) <- seqs alignment <- AlignSeqs(DNAStringSet(seqs), anchor=NA,verbose=FALSE)
- Y con ese alineamiento inferimos un árbol de partida o starting tree para inicializar la búsqueda por ML. También ajustamos un modelo de sustitución nucleotídica para parametrizar la tasa de cambio de un nucleótido a otro y asó poder inferir correctamente el largo de las ramas y la topología del árbol.
phangAlign <- phyDat(as(alignment, "matrix"), type="DNA") dm <- dist.ml(phangAlign) treeNJ <- NJ(dm) fit = pml(treeNJ, data=phangAlign) fitGTR <- update(fit, k=4, inv=0.2) fitGTR <- optim.pml(fitGTR, model="GTR", optInv=TRUE, optGamma=TRUE, rearrangement = "stochastic", control = pml.control(trace = 0))detach("package:phangorn", unload=TRUE)
Ahora, tenemos todos los ingredientes para formar un objeto de R que contenga todo lo que nos importa en un experimento metagenómico, i.e., una tabla de cuentas que indica el número de reads por seceuncia del 16S rRNA gene, una tabla con el linaje taxonómico de esas secuencias, un árbol filogenético que relaciona esas secuencias entre sí, y finalmente, una tabla con variables asociadas a nuestras muestras, también llamada metadata.
Descarga la metadata AQUÍ.
Con estos elementos procedemos a generar un objeto phyloseq:
samdf <- read.csv("~/Dropbox/CastroLab_database/workshops_data/metadata.csv", header=TRUE, row.names = 1)
rownames(seqtabNoC) %in% rownames(samdf)
all(rownames(seqtabAll) %in% samdf$run)
ps <- phyloseq(otu_table(seqtabNoC, taxa_are_rows=FALSE),
sample_data(samdf),
tax_table(taxTab),
phy_tree(fitGTR$tree))
ps <- prune_samples(sample_names(ps) != "Mock", ps) # Remover potenciales muestras sintéticas
ps
Y el producto final del análisis por DADA2 es entonces un objeto phyloseq:
Slot Descripción Resultado
otu_table() OTU Table: [ 1476 taxa and 98 samples ]
sample_data() Sample Data: [ 98 samples by 13 sample variables ]
tax_table() Taxonomy Table: [ 1476 taxa by 6 taxonomic ranks ]
phy_tree() Phylogenetic Tree: [ 1476 tips and 1474 internal nodes ]
Introducción a phyloseq y a análisis de diversidad
1 Instalación de paquetes de R
Los siguientes 3 scripts te mostrarán una manera eficiente de instalar y cargar la lista de paquetes según su repositorio de origen, ya que cada repositorio tiene su propia función para instlar sus paquetes.
- Primero, enlistar los paquetes necesarios en diferentes vectores dependiendo de su repositorio de origen.
## Repositorio CRAN
cran_packages <- c("bookdown", "knitr", "tidyverse", "plyr", "grid", "gridExtra", "kableExtra", "xtable", "ggpubr")
## Repositorio Bioconductor
bioc_packages <- c("phyloseq", "dada2", "DECIPHER", "phangorn", "ggpubr", "BiocManager","DESeq2", "microbiome", "philr")
## Repositorio GitHub
git_source <- c("twbattaglia/btools", "gmteunisse/Fantaxtic", "MadsAlbertsen/ampvis2", "opisthokonta/tsnemicrobiota") # fuente/nombre del paquete git_packages <- c("btools", "fantaxtic", "ampvis2", "tsnemicrobiota") # nombre del paquete
- Segundo, instalar los paquetes definidos arriba usando la función correspondiente a cada repositorio.
# Instalar paquetes CRAN
.inst <- cran_packages %in% installed.packages()
if(any(!.inst)) { install.packages(cran_packages[!.inst]) }
# Intalar paquetes BioConductor if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager") .inst <- bioc_packages %in%
installed.packages() if(any(!.inst)) { BiocManager::install(bioc_packages[!.inst]) }
# Instalar paquetes GitHub .inst <- git_source %in% installed.packages() if(any(!.inst)) { devtools::install_github(git_source[!.inst]) }
- Tercero, cargar los paquetes requeridos a la sesión actual de R.
# Cargar paquetes sapply(c(cran_packages, bioc_packages, git_packages), require, character.only = TRUE)
El paso de instalación de paquetes es necesario solamente una vez. Excepto si se quiere actualizar la versión del paquete, o bien, R ha sido desinstalado e instalado nuevamente o actualizado su versión.
Si ya tienes los paquetes instalados en tu computadora, sólo necesitas enlistar (1er script) y cargar (3er script) los paquetes. También, puedes cargarlos a la sesión actual de R usando la función library(), así:
# Cargar paquetes
library(tidyverse)
library(plyr)
library(grid)
library(gridExtra)
library(kableExtr)
library(xtable)
library(ggpubr)
library(phyloseq)
library(dada2)
library(DECIPHER)
library(phangorn)
library(ggpubr)
library(BiocManager)
library(DESeq2)
library(microbiome)
library(philr)
library(btools)
library(fantaxtic)
library(ampvis2)
library(tsnemicrobiota)
2 Introducción a phyloseq
Phyloseq es un paquete de Bioconductor (Open Source Software For Bioinformatics) para la manipulación y análisis de datos metagenómicos generados por metodologías de secuenciación de alto rendimiento. phyloseq es una herramienta para importar, guardar, analizar y visualizar éste tipo de datos después de haber sido procesados inicialmente, e.g., ensamblaje de novo, ASVs u OTUs (clustered), incluyendo otros importantes datos asociados (si están disponibles): tabla de observaciones asociadas a cada muestra (e.g., especie, localización geográfica, temperatura, etc.), conocida como sample data o metadata, árbol filogenético, e identificación taxonómica de cada OTU. La estructura del paquete phyloseq consiste en una serie de funciones de acceso y de proceso de objetos phyloseq. Estos objetos están compuestos de cuatro componentes que almacenan las cuentas de reads, la metadata, la taxonomía y el árbol filogenético. El paquete también provee una serie de herramientas para importar datos de otros programas. El siguiente diagrama muestra la estructura completa de phyloseq.

Si no tienes el objeto phyloseq ps generado por DADA2 puedes descargarlo haciendo clic: AQUÍ
En la terminal de R cargamos el objeto phyloseq:
# Leémos el objeto phyloseq del análisis por DADA2 readRDS("ps.RDS") -> psd psd
## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 1476 taxa and 98 samples ]
## sample_data() Sample Data: [ 98 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 1476 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 1476 tips and 1474 internal nodes ]
2.1 Control de calidad del análisis de 16S
Lo primero que podemos mirar es la prevalencia de las features taxonómicas.
- Primero creamos un data frame con los valores de prevalencia, luego les agregamos la taxonomía y graficamos.
# Computamos prevalencia para cada feature y la guardamos en un data frame prevdf = apply(X = otu_table(psd),
MARGIN = ifelse(taxa_are_rows(psd), yes = 1, no = 2),
FUN = function(x){sum(x > 0)})
# Le agregamos la taxonomía
prevdf = data.frame(Prevalence = prevdf,
TotalAbundance = taxa_sums(psd),
tax_table(psd))
plyr::ddply(prevdf, "Phylum", function(df1){cbind(mean(df1$Prevalence),sum(df1$Prevalence))}) -> dfprev
kable(dfprev)
Phylum
Acidobacteria 3.800000 38
Actinobacteria 5.0416673 63
Bacteroidetes5.7193882242BRC11.0000001Chloroflexi1.75000021Ciliophora3.0000009Cyanobacteria4.223881283Deinococcus-Thermus1.3333334Epsilonbacteraeota2.76000069Euryarchaeota3.71428626Firmicutes4.685315670Fusobacteria3.50000070Gemmatimonadetes1.0000002Kiritimatiellaeota1.0000001Lentisphaerae1.2500005Marinimicrobia_(SAR406_clade)3.5000007Nanoarchaeaeota1.0000001Nitrospinae1.0000001Ochrophyta1.0000001Patescibacteria5.473684312Planctomycetes3.235294110Proteobacteria8.2338404331Schekmanbacteria1.0000001Spirochaetes1.5000003Tenericutes6.08333373Thaumarchaeota4.00000012Verrucomicrobia4.814815130NA6.425532302
Al examinar la tabla, es evidente que algunos Phylum aunque presentes, están muy poco representados. La columna 1 representa la media de read counts para ese Phylum, mientras que la columna 2 representa la suma. Por ejemplo, grupos como BRC1, Kiritimatiellaeota, y Nanoarchaeaeota están representados solamente por 1 read. Es muy riesgoso mantener estos grupos taxonómicos en el análisis ya que pueden corresponder a falsos positivos.
- Para filtrarlos, generamos un vector con todas las taxa que queremos filtrar.
# Definimos taxa a filtrar filterPhyla = c("BRC1", "Deinococcus-Thermus", "Gemmatimonadetes", "Kiritimatiellaeota", "Nanoarchaeaeota", "Ochrophyta", "Schekmanbacteria", "Ciliophora", "Spirochaetes", NA)
# Procedemos a filtrar
(psd1 = subset_taxa(psd, !Phylum %in% filterPhyla))
## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 1414 taxa and 98 samples ]
## sample_data() Sample Data: [ 98 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 1414 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 1414 tips and 1412 internal nodes ]
# Además aprovechamos a remover taxa que no corresponde a microorganismos como cloroplastos, mitocondrias y otros
filterPhyla2 <- c("Chloroplast", "Mitochondria", "Eukaryota")
psd1 <- subset_taxa(psd1, !Kingdom %in% filterPhyla2)
psd1 <- subset_taxa(psd1, !Phylum %in% filterPhyla2)
psd1 <- subset_taxa(psd1, !Class %in% filterPhyla2)
psd1 <- subset_taxa(psd1, !Order %in% filterPhyla2)
psd1 <- subset_taxa(psd1, !Family %in% filterPhyla2)
psd1 <- subset_taxa(psd1, !Genus %in% filterPhyla2)
- Además del filtrado que acabamos de realizar, existen otros tipos de filtrado que tienen que ver con la media de read counts por taxa, con la distribución de éstas, y con filtrar muestras bajo un número mínimo de reads.
# Filtramos taxa de acuerdo a un umbral de número medio de _read counts_, en este caso 1e-5 psd2 <- filter_taxa(psd1, function(x) mean(x) > 1e-5, TRUE)
# También podemos remover taxa que no se observe más de X veces en al menos 10% de las muestras psd3 <- filter_taxa(psd2, function(x) sum(x > 2) > (0.1*length(x)), TRUE)
# Y finalmente filtrar muestras con menos de 1000 reads psd4 = prune_samples(sample_sums(psd3) > 1000, psd3)
psd4
## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 136 taxa and 87 samples ]
## sample_data() Sample Data: [ 87 samples by 13 sample variables ]
## tax_table() Taxonomy Table: [ 136 taxa by 6 taxonomic ranks ]
## phy_tree() Phylogenetic Tree: [ 136 tips and 135 internal nodes ]
- Otra forma de filtrar taxa de baja prevalencia es estableciendo un umbral y luego visulizar el efecto de manera grafica.
# Seleccionamos las taxa de interés prevdf1 = subset(prevdf, Phylum %in% get_taxa_unique(psd4, "Phylum")) ggplot(prevdf1, aes(TotalAbundance, Prevalence / nsamples(psd),color=Phylum)) +
# Agregamos una línea para nuestro umbral
geom_hline(yintercept = 0.05, alpha = 0.5, linetype = 2) + geom_point(size = 2, alpha = 0.7) +
scale_x_log10() + xlab("Total Abundance") + ylab("Prevalence [Frac. Samples]") +
facet_wrap(~Phylum) + theme(legend.position="none")

# Definimos el umbral de prevalencia a un 5% (prevalenceThreshold = 0.05 * nsamples(psd4))
## [1] 4.35
# Execute prevalence filter, using `prune_taxa()` function keepTaxa = rownames(prevdf1)[(prevdf1$Prevalence >= prevalenceThreshold)] (psd5 = prune_taxa(keepTaxa, psd4))
## phyloseq-class experiment-level object ## otu_table() OTU Table: [ 136 taxa and 87 samples ] ## sample_data() Sample Data: [ 87 samples by 13 sample variables ] ## tax_table() Taxonomy Table: [ 136 taxa by 6 taxonomic ranks ] ## phy_tree() Phylogenetic Tree: [ 136 tips and 135 internal nodes ]
DADA2 usa como nombre de las taxa la secuencia o ASV asociada a un taxon determinado. Esto es conveniente cuando nos interesa la secuencia en nuestros análisis, sin embargo en este práctico solamente vamos a trabajar a nivel de comunidad.
- Por esto, vamos a reemplazar esos nombres con códigos correlativos, lo cual va a hacer las visualizaciones posteriores más entendibles.
# Reemplazamos las secuencias por un nombre genérico taxa_names(psd5) <- paste0("ASV", seq(ntaxa(psd5)))
- Con nuestro objeto phyloseq ya filtrado y listo para usar, podemos gráficar la distribución de read counts por número de muestra de forma de tener una idea sobre la distribución de éstas.
sample_sum_df <- data.frame(sum = sample_sums(psd5))
ggplot(sample_sum_df, aes(x = sum)) +
geom_histogram(color = "black", fill = "grey", binwidth = 2500) +
ggtitle("Distribution of sample sequencing depth") +
xlab("Read counts") +
theme(axis.title.y = element_blank())
- Finalmente, calculamos curvas de rarefacción para cada muestra, de manera tal que podamos determinar si la profundidad de secuenciación fue sufuciente o si tal vez necesitemos secuenciar más. En otras palabras, este análisis nos permitiría averiguar si al secuenciar más observaríamos más OTUs o ASVs.
# Primero cargamos algunos scripts de manera remota scripts <- c("graphical_methods.R", "tree_methods.R",
"plot_merged_trees.R",
"specificity_methods.R",
"ternary_plot.R",
"richness.R", "edgePCA.R", "copy_number_correction.R", "import_frogs.R", "prevalence.R", "compute_niche.R")
urls <- paste0("https://raw.githubusercontent.com/mahendra-mariadassou/phyloseq-extended/master/R/", scripts)
for (url in urls)
{ source(url) }
# Y graficamos
p <- ggrare(psd5, step = 100, color = "species", label = "sample_ID", se = TRUE)
(p <- p + facet_wrap(~species))
Los gráficos están separados por especies de ballena, azul (Balaenoptera musculus), fin (Balaenoptera physalus), franca (Eubalaena australis), y jorobada (Megaptera novaeangliae) y muestran la cantidad de Taxa (riqueza o diversidad alfa) en función del tamaño muestreal o número de reads. Podemos ver que la mayoría de las muestras de ballena azul y jorobada alcanzan un plateau. Esto significa que el retorno en la inversión disminuye si seguimos secuenciando, o de otra forma, que ya hemos muestreado toda la diversidad de esa muestra. Al contrario, algunas muestras de ballena fin no alcanzaron el plateau, lo cual implica que la diversidad alfa estaría subestimada.
2.2 Estructura y manipulación de un objeto phyloseq
Muchas veces queremos analizar un sub conjunto de las muestras en nuestro objeto phyloseq, o bien, queremos seleccionar ciertos grupos taxonómicos para análisis posteriores. phyloseq nos permite hacer todo tipo de filtros para llevar esto a cabo. Veamos dónde se almacena la información en phyloseq.
- Primero la tabla de cuentas.
La tabla de cuentas relaciona el nombre de las taxa con las muestras y con el número de reads mapeadas en contra de ellas. Acá el número de reads es directamente proporcional al número de veces que se observa un taxon.
- El otro componente importante es la tabla de taxonomía.
- Kingdom Phylum Class Order Family Genus
ASV1 BacteriaProteobacteriaGammaproteobacteriaCardiobacterialesCardiobacteriaceaeNAASV2BacteriaBacteroidetesBacteroidiaFlavobacterialesFlavobacteriaceaeNAASV3BacteriaProteobacteriaGammaproteobacteriaPseudomonadalesMoraxellaceaeNAASV4BacteriaProteobacteriaGammaproteobacteriaNANANAASV5BacteriaProteobacteriaGammaproteobacteriaXanthomonadalesXanthomonadaceaeStenotrophomonasASV6BacteriaProteobacteriaGammaproteobacteriaPseudomonadalesMoraxellaceaeMoraxella
Textos y glosario que utilizamos
https://www.nature.com/subjects/
https://doegenomestolife.org/glossary/
TIPOS DE PLATAFORMAS MÁS IMPORTANTE DE MICROARREGLOS
https://www.agilent.com/labs/features/2011_101_microarray.html
https://www.illumina.com/techniques/microarrays.html
https://www.affymetrix.com/about_affymetrix/outreach/educator/microarray_curricula.affx
MICROARREGLOS DE SEGUNDA: 500 Arrays.
https://www.microarrays.com/dna-arrays.php
NUESTRO ENVÍO DE MATERIAL GENÉTICO POR FEDEX-CHILE
Conceptos y preguntas básicas de Biotecnología Celular y Molecular
The lower the E-value, or the closer it is to zero, the more "significant" the match is. However, keep in mind that virtually identical short alignments have relatively high E values. This is because the calculation of the E value takes into account the length of the query sequence. These high E values make sense because shorter sequences have a higher probability of occurring in the database purely by chance. For more details please see the calculations in the BLAST Course. The Expect value can also be used as a convenient way to create a significance threshold for reporting results. You can change the Expect value threshold on most BLAST search pages. When the Expect value is increased from the default value of 10, a larger list with more low-scoring hits can be reported.
3.- Análisis del paper "Dysregulation of mitotic machinery genes procedes genome inestability during spontaneous pre-malignant transformation of mouse ovarian surface epithelial cells" (Pasantía doctoral, Facultad de Medicina, Universidad de Chile).
¿Porque es importante la superficie epitelial ovárica en un proceso ovulatorio para estudio de cáncer?
En estudios sobre la superficie ovárica hay muchos paper. Se piensa que esas células dan origen a los tumores ováricos. Hace unos años atrás se empezó a dilucidar que el origen no necesariamente podía ser ovárico, si no podría ser del oviducto o trompa de Falopio, entonces actualmente se despertó el interés por el estudio del origen real del cáncer, aunque suena un poco raro hablar de cáncer ovárico y decir que su origen está en el oviducto. Por tanto, actualmente se disputa por el origen real de éste cáncer. Este estudio está enfocado a estudios de la superficie ovárica de ratón y lo que se hace es un sistema "in vitro". En la Fig. 1 del paper, se muestra un esquema donde se tomaron los extractos de ADN y ARN en distintas etapas o ciclos de división celular, dejaron que el cultivo se dividiera progresivamente tomando extractos de ADN y ARN en distintas etapas. Por tanto, éste es un estudio "in vitro", no estudia transformaciones en el órgano, tiene su limitación.
El cáncer es una transformación de tipo celular, pero hay obviamente una interacción con el estroma, el sistema inmune y varios tipos de células que son importantes en el desarrollo de un tumor eso sería la limitación del trabajo, que es un sistema "in vitro". Aún así es posible extrapolar hasta la enfermedad real en humanos. En la figura 1, se hizo la toma de ARN y ADN total y se hace un microarreglo para ver que ésta presente en esa linea celular, usando como referencia el genoma de ratón Mus musculus. En este estudio para el microarreglo se tomaron muestras de ARN de referencia (una muestra de ratón recién nacido completo) y ADN de referencia. Se hicieron experimentos separados de ARN total de células que están siendo transformadas "in vitro", contra un ARN de referencia.
¿Cuál es lo común en un organismo multicelular?
El ARN no es común, el ADN es común. Por tanto, si yo quiero tener un ARN de referencia, debo tener en cuenta, que en la mayoría de los tejidos posibles en teoría, en el mismo microarreglo se observe todos los spots prendidos, teniendo una cobertura total de todos los mensajeros. Los ratones recién nacidos tienen una piel transparente sin pelos, observándose los órganos, son bastante delicados, donde la idea es tener representado todos los mensajeros. En cambio, si se estudia al hígado u otro órgano, la mayoría podría estar apagado, por eso se estudia la mezcla de todos esos genes. También hay empresas que ofrecen ARN de referencia que obtienen de 10 a 15 líneas celulares típicos de tejidos de hígado, riñón, musculo, cerebro, etc. venden el ARN total, con ese ARN de referencia estoy comparando todos los ARN de los respectivos pasajes. En el caso del ADN, se compara el ADN de sangre de ratón adulto normal, es decir el genoma normal, frente al genoma de estas células que están siendo transformadas que es lo que se quiere observar al analizar ADNc, para lo cual es necesario observar un arreglo cromosómico que no se ven en el microarreglo debido a que en la mayoria de los tipo de cáncer se observa ANEUPLOIDIAS llamadas AMPLIFICACIONES o DELECCIÓN de cromosomas completos, segmentos de diferentes cromosomas, siendo un proceso llamado INESTABILIDAD GENÓMICA.
En cuanto al uso de la palabra "espontaneo" en el paper, es una de las gracias de este modelo, en la cual no se usó ningún agente carcinógénico, ni xenobióticos, ni virus, siendo un proceso totalmente espontaneo, el cual se había discutido que ocurría en células de rata, de humano del mismo origen, siendo una clase de proceso espontáneo, que es producida por la repetición sucesiva de pasajes "IN VITRO". La idea es tener una herramienta que detecte precozmente el cáncer. Rosse hizo un trabajo similar donde las células sobre el pasaje 20 podían ser capaces de inducir tumores, y en ese aspecto de cáncer ovárico se parece mucho a lo que ocurre en el caso de humano. Se usaron células inducidas de esta forma para inducir tumores en ratones viejos y la diseminación de tumores era exactamente igual a como se describe en el caso de tumores de mujeres. Las células observadas sobre el pasaje 20, y en realidad se observa con tumores ováricos intactos y líneas celulares derivadas de tumores ováricos, es decir tratando de generar un tumor en un animal y que el tumor sea desarrollado en forma espontánea es algo bastante difícil. La incidencia de este cáncer es bastante baja. Para reproducir esto en un animal se debe tener varios miles de ratones y quizás observar en cual de ellas se puede formar un tumor espontáneo no es fácil de hacerlo. Estas líneas celulares MOSE han sido usadas en bastantes modelos haciendo una búsqueda en PUBMED se han estudiado diversos procesos de la tumorigénesis ovárica siempre en animales. Una de las gracias de esta célula es que nos permite usar animales transgénicos y no animales inmunodeprimidos normales.
En la Figura 2 se observan las barras amarillas de ARN y barras azules de ADN, graficándose los clones que arrojan la expresión diferencial mediante niveles diferenciales, En el caso de expresión, cuando se está analizando el ARN, en el otro caso, son niveles diferenciales de ADN.
¿Como se obtuvieron estos valores que están graficados en la figura 2.A?
Usaron el p2 como el basal para el caso del ARN. Hay formas de hacer, lo que se llama raspados de ovario, la cantidad de células que se obtenía eran muy raras y variables, al final se usó como células normales en el pasaje 2 haciendo el t-test de todo el resto de pasajes frente al pasaje 2, el test limma también permite comparar dos grupos, de ahí salieron todos los resultados que se observa en la figura 2A.
Durante el 1er pasaje hasta el pasaje 14, se hizo un análisis breve de los genes en el pasaje 5. En el pasaje 14 los clones son de 280 y 50. En la fig. 2.A se observa 245 genes que se dividen en 101 genes upregulated y 80 genes downregulated. Por tanto, hay genes repetidos, siendo la principal razón y hay otros genes que no tienen anotación que son desconocidos, siendo la mayoria.
Esta biblioteca que es NIA-15K cADN, en el pasaje 14 hay una cantidad significativa del número de genes expresados diferencialmente donde el ADN es significativamente bajo, en el pasaje 18 aumenta el ADN. Por tanto, lo que está en la figura 2B me dice que es lo que se mantiene o no se mantiene en el curso del proceso mediante el diagrama de Venn. Prácticamente se observan los 3 grupos donde hay coincidencias, entonces las coincidencias son bastante pocas a excepción del pasaje 23 y 28 donde tengo 52 y 69 genes. siendo más amplio en el número de genes. En el pasaje 18 no se observan ningún gen exclusivo de ese pasaje. La tabla 1 es un resumen que se hizo un análisis de GENE ONTOLOGY de todos los genes UP y DOWN regulated del pasaje 14, donde la lista de UP son 53 de 101, es decir cubre un poco más de la mitad y DOWN regulated cubre menos de la mitad, hay una parte de los genes donde no aparecen en ninguna función de GENE ONTOLOGY, para esto se uso el programa Web Gestalt, siendo rankeado en enriquecimiento. Por tanto, lo que esta en la tabla 1 es una recopilación bibliográfica.
En la figura 3A se observa una madeja de
interacciones, marcándose todos los genes complejos de la tabla 1, Por ejemplo
centralspindlin está colocado en la red de interacciones. En la figura 4, se
observa los datos de la plataforma ARRAYS CGH siendo necesario ponerlo en el
contexto cromosomal, donde en la figura 4B se observa de que hacia arriba hay
una amplificación y abajo se observa la delección. En la figura 4.A en la cual
cada una de las lineas centrales que están adyacente a la derecha seria la
amplificación y el de la izquierda seria la delección, donde en los cromosomas
en las líneas 1,2 y 3 la tendencia va por la línea central, deduciendo que ahí
no hay aberraciones cromosómicas, en cambio en los cromosomas 10 y 17 se
observan amplificaciones; en el cromosoma 19 se observan una delección.
4.-¿Como hacer microarreglos?
Tengo un ARN de referencia y un ARN de interés y se marca con dos fluorofóros a través de una transcripción reversa con fluorofóros Cy3 y Cy5, todos los microarreglos son de dos canales, un canal verde y un canal rojo, son seudocolores de fluorescencia, colores de fantasía, siendo esos los fluorofóros que se usaron desde un principio que se desarrolló la tecnología y a principios de los años 90. Entonces se desarrolló esa tecnología de dos canales, donde se marca separadamente y después se mezcla esas dos muestras y se cohíbrida sobre el microarreglo.
5.- ¿Que es un microarreglo?
Es un soporte rígido, típicamente un portaobjeto común y corriente, sobre el cual están depositadas las sondas, en este caso son clones de ADNc, en el fondo son sondas. La primera fase del desarrollo de la tecnología desde el 1990 hasta el 2010, se usó bastantes clones de ADNc y progresivamente las tecnología fueron cambiando, fueron desarrollándose los microarreglos que tenían sondas de diferentes longitudes, las típicas robustas oscilan entre 40 y 70 nucleótidos, eso es lo que hoy en día se puede encontrar comercialmente, cohibridando muestras marcadas sobre esta superficie que tiene depositada o unida covalentemente cada una de estas sondas como los genes tenga el genoma, es decir, si se desea tener un microarreglo completo se debería tener por lo menos, en el caso de humanos, unas 20500 sondas depositadas cada una, en un lugar distinto en el espacio del microarreglo. Actualmente, también se han depositado sondas para medir genes no codificantes.
En realidad se quisiera medir el
transcrito completo en un formato de microarreglos más o menos de unas 40000
sondas distintas. En caso de microARN es un poco problemático, probablemente el
método de marcaje será distinto, porque la secuencia con la cual estamos
trabajando, mientras más chica o pequeña sea, la posibilidad que se una inespecíficamente
a una macromolécula de ADN compleja es mayor, entonces debe existir
microarreglos de microARN, actualmente debe de existir 2000 más o menos. La
tecnología de microarreglos se puede ir miniaturizando más, de tal manera en
ese mismo espacio donde se colocaba un microarreglo completo que nos sirve
para comparar una muestra con su referencia; ahora las compañías están
colocando hasta 8 microarreglos en sentido horizontal, la gracia de estos
microarreglos, es que esta sonda esta repetida en diferentes lugares al menos
unas 30 veces más, haciendo un escáner de mucha mayor resolución. Actualmente
existen tres tipos de plataformas de microarreglos: ilumina, Agilent y
Affymetrix.
6.- ¿Como hacer microarreglos con ADN?
El proceso es esencialmente lo mismo, pero tiene algunos problemas ¿Cual sería el problema entonces? El ADN tiene intrones (específicos). Si tratamos de hibridar el ADN del genoma completo con las sondas de secuencia de ADN codificante nos daría una baja resolución. A pesar de ser un problema también nos permitiría obtener algunos datos.
7.- ¿Como están distribuidos los genes del genoma?
Los genes dentro del genoma son como islas en medio del desierto del genoma del ADN no codificante. Por tanto se tendrá una información sesgada si se hibrida ADN contra un sets de sondas de ADN solamente de genes codificantes, siendo el problema que se presenta. Es decir, una baja resolución que ofrece el microarreglo pensado para hacer análisis de expresión de ADN, aún así se puede obtener algunas conclusiones del genoma.
8.- ¿Cuál es el tamaño completo del genoma humano?
3200 Megabases entonces si se divide el tamaño entre el número de sondas, nos dará una baja resolución. Para lo cual se hace Arrays CGH que es la hibridación genómica comparativa, el cual consiste en diseñar sondas que cubre en forma homogénea el genoma sea que haya regiones codificantes o no. Hay regiones que tienen más densidad de genes que otras, entonces un Arrays CGH actual tiene una cobertura homogénea.
9.-¿Que se debe tener en cuenta para el secuenciamiento?
Se puede mandar a secuenciar a Macrogen(Korea) o BGI(China) , la empresa hace todo el experimento como el marcaje y el dispositivo, haciendo un control de calidad al ARN a través de una maquina byoanalizer, el cual funciona por electroforesis capilar, observándose la abundancia de ARN ribosomal midiendo la integridad del ARN, éste debe estar puro e intacto, sin ningún rastro de degradación, siempre una muestra original es un ARN pasando siempre a ADNc y de hecho para la plataforma Ilumina se sintetiza un ARN complementario. Se usa un transcripción "in vitro" primero se hace la primera hebra y luego se genera un templado para una polimerasa y finalmente se marca con fluorofóro. Si el ARN llega en mal estado a la empresa de secuenciamiento no sirve para el propósito esperado.
10.- Print tip Loess normalization:
Es una normalización que requiere generar los datos en este formato, sirviendo para homogenizar la distribución de los datos. Es decir, si se tiene un microarreglo con una muestra se debe analizar la distribución de los datos. En el formato de dos canales, los puntitos amarillos no nos sirven.
En el microarreglo se marca con dos fluorofóros,
se mezcla, se híbrida y luego se pasa por el escáner de fluorescencia, donde
aquellos mensajeros que están en el mismo nivel, aparecen con una señal
amarilla que significa que hay igual intensidad de color, lo que interesa son
los spots verdes y rojos, que están más representados en una muestra que la otra.
Entonces en la práctica, si se analiza la distribución de un microarreglo,
observamos el color amarillo, rojo y verde. La normalización lo que puede hacer
es centrar todo el amarillo puro por decirlo de otra forma que es en buenas
cuentas log2 que es lo que se usa para analizar razones de expresión. Lo que
hace la técnica de dos canales y también la de un canal solo que se hace IN
SILICO, directamente se analizaba la razon de (rojo)x(verde), (muestra de interés)x(muestra
de referencia) y mientras más alto es ese número, significa que ese gen está
más representado por la muestra que se marcó. Por tanto, es necesario realizar
la normalización denominada "Print tip Loess
normalization". ¿Porque se genera estas diferencias? Esta
distribución rara significa que todos mis spots lo estoy viendo globalmente más
rojizo o más verdoso, dependiendo del lado que se esté eligiendo, lo cual me induciría
a pensar que a lo mejor hay un problema con el marcaje rojo o que el fluoróforo
se está degradando o el ARN tenía una impureza o al ARN que se marcó con ese
fluoróforo algo le paso frente al otro que anduvo bien, por eso se genera ese
tipo de imagen lo que en la práctica ocurre. Lo que se ve en libros o internet
esas imágenes que parecen semáforos que se ven es el experimento perfecto, que
a veces es posible tenerlo, pero cuando ya no se puede, ya no se puede hacer
otra hibridización porque ya se habría gastado en muestra, las enzimas, los
fluoróforos que son carísimos, el vidrio, no se puede perder, siendo la forma
de rescatar estos experimentos que son un poco defectuosos desde el punto de
vista técnico, donde la normalización es nada más que técnica, alterando todas
las razones de todos los spots que se midió en el microarreglo sin saber cuál
spots esta alterado o no. Es una normalización completa al sets de datos
completos, no hay ningún sesgo porque a veces uno entiende que esta alterando
los datos, forzando numéricamente a que la distribución sea homogénea en todo
el sets de experimentos que estoy haciendo, a eso se refiere el LOESS. En
el caso de los microarreglos de un canal, la evidencia hace una normalización
por cuartiles, que no es otra cosa que una normalización por ranking, donde se
puede tener exactamente el mismo fenómeno, las distribuciones podrían tener un
máximo diferente, en este caso el rankeo el que tiene mayor fluorescencia lo
igualo a otro y así los voy igualando haciendo que los dos ocupen el mismo
orden en un ranking, siendo la normalización por cuantiles.
11.-TRANSCRIPTÓMICA:
Un organismo que no está secuenciado, no se conoce su genoma. Por tanto, no dará mucha información, entonces una alternativa correcta es el ARNseq, de ahí a que independientemente se conozca o no el genoma del organismo, lo que se está expresando; eventualmente se puede encontrar todos los ARN que codifica para algo, ya que es un eucarionte, donde los genes tendrían que ser monocistrónicos. En cambio un gen que puede codificar para un ARNm o puede codificar para más de un ARNm como un operon, siendo propio de los procariontes. Hay técnicas para hacer ARNseq. Si el ARN tiene 5´cap en un extremo y un PoliA en el otro extremo, se trata de un ARNm, estaríamos aislando el ARNm para su secuenciación, independientemente de que se conozca o no se conozca.
Después de la secuenciación, lo que se puede evaluar, es la abundancia de los transcritos. Básicamente lo que se hace en un ARNseq, primero se extrae el ARN del organismo, el ARNm en el caso de un eucarionte, siendo fácil, luego se secuencia. Al secuenciar el ARNm, secuenciamos todo el ARNm, dependiendo de la técnica que se va ocupar, de acuerdo al kit de secuenciación que se va ocupar nos aseguramos de que casi el 90 o 95% de todos los ARNm, los pillemos en esta secuenciación, existiendo ARN que no alcanzo a secuenciarse, siendo muy poco; eso depende de la profundidad de secuenciación, el cual depende de un sistema de secuenciación, que involucra eventualmente capturar las secuencias de muchos ARN, casi la totalidad en algunas condiciones. Cuando se trata de un organismo que no tiene muchos tejidos, éste no tiene expresión diferencial, órganos diferentes, es mucho más fácil.
Al capturar el ARNm de acuerdo a los tratamientos a diferentes tiempos, se puede cuantificar la cantidad de transcritos teniendo aproximaciones estadísticas, el cual sirve para evaluar la expresión relativa de un tiempo con respecto a otro, haciendo un análisis estadístico que relacione el tratamiento con el tiempo, con un test variado.
¿Que sacamos con eso? Por ejemplo se puede tener una cantidad de transcritos con respecto al tiempo 1, el tiempo 0 con respecto al tiempo 2 y así sucesivamente.
t0-t1
t1-t2
t0-t2
t1-t3
t0-t3
t2-t3
Podemos tener todas las combinaciones, entonces de acuerdo a la cantidad de transcritos, por decir el gen "x" puedes estar 3 veces expresado en el tiempo 1(t1) con respecto al tiempo 0(t0), ó el gen "x" en el t2 disminuye el doble al tiempo 0, ...... , total pueden darse todas las opciones que uno pueda encontrar, entonces teniendo todos estos perfiles de expresión, podemos evaluar que genes nos sirven para el patrón de expresión, todo eso se normaliza.
¿Estaremos familiarizados con el ARNseq?:
El ARNseq es una libreria de ARNm, entonces al tener ARN, éste se fragmenta en pedacitos que son los ARNm, al final obtendremos fragmentos básicos de 150 pares de bases de tamaño máximo, entonces estos pequeños fragmentos lo voy a empalmar por homología de secuencia, dando lugar a un CONTIG que son secuencias largas, donde puede estar contenido un trascrito de forma completa, entonces los pequeños fragmentos que son los que se secuenciaron se denominan READS. Éstos dependen de varias cosas, como de kits que se ocuparon, así como del largo del gen. Si el gen es muy grande o muy corto. Eventualmente en genes muy grandes, tendremos una mayor cantidad de READS, independientemente que este gen haya estado más sobreexpresado que otro, porque al final, la cantidad de READS nos tiene que dar una luz de cómo estaba expresado la abundancia del transcrito con respecto a cada gen, pero imaginemos que tenemos un transcrito muy grande(100 ARNm) con respecto a un trascrito muy corto(3ARNm), yo al fragmentar genero los READS, entonces en los transcritos grandes es donde tenemos más READS, porque es más largo, al obtener los READS, debemos evaluar la calidad de secuenciación, siendo parámetros que se deben normalizar por el conteo total de READS que obtuvimos por tamaño del genoma, en el caso que se haya secuenciado, existiendo varias técnicas estadísticas para normalizar, con esos READS normalizados, podemos mapear en un genoma de referencia, siendo el genoma una secuencia larga donde están representados todos los genes del organismo. Entonces el gen 1 tiene cierta cantidad de READS, el gen 2 puede tener otra cantidad de READS y asi sucesivamente, Entonces podemos decir que un gen que tiene mas READS, es probable que este más sobreexpresado que otro gen que tiene menos READS. Eso es cuando tenemos el genoma de referencia, pero si no tenemos el genoma de referencia también lo podemos hacer. Podemos hacer una toma de referencia. Es decir, todos estos READS empalmarlos como si fuera un genoma, en vez de tener mi GENOMA DE REFERENCIA voy a tener mi TRANSCRIPTOMA "Todos mis CONTIG van a representar a mis transcritos mensajeros para representar al gen", donde se puede realizar un mapeo.
¿Cuál es la ventaja con respecto a una micromatriz?
En una micromatriz debo conocer el genoma para poder ver su expresión, en cambio en el TRANSCRIPTOMA, se puede encontrar la expresión de cosas que no se sabía, no se conocía que estaban ahí, Entonces, mediante esta técnica, se puede abarcar toda la cantidad de ARNm que existen en el organismo. Y con el análisis de datos que genes están sobreexpresandose de forma ciclica. Ahora se puede hacer una anotación, porque se genero el transcriptoma de referencia se va empalmando todos los READS, dándo secuencias más largas, empalmando por "homología de secuencia" , ahora si se las quiere en una base de datos, hago un BLAST, este programa me va decir que este gen eventualmente se parece a una actina o a una ciclina, o a una Cdk, etc. lo que nos interesa, teniendo una cierta cantidad de genes que los puedo ir mirando. Los principios de secuenciación del método de Singer, se secuencia mediante el ensamble de secuencias cortas, que van a empalmar y llegamos a la secuenciación masiva, entonces los READS que son los que se secuenciaron se normalizan.
Para el secuenciamiento, los ´programas bioinformáticos que se utilizan, no son sencillos de aprenderlo. Los programas se realizan con Linux, no corren en windows, necesitando una gran cantidad de memoria RAM. Es decir, una alta cantidad de almacenaje, se deben ensamblar genomas grandes; las laptop de uso diario me podrían servir para ensamblar genomas pequeños pero no genomas grandes dependiendo de la cantidad de READS, Por cada 100000 READS requiero usar 1 Gigabyte(GB) de RAM, si tuviera 400000000 de READS se requiere mayor memoria RAM.
El Trinity es el programa que permite ensamblar el genoma. Por ejemplo en una lista de 138000 CONTIG siendo el transcriptoma de referencia, esos CONTIG provienen de READS que empalmaron. Esa sumatoria de CONTIG, sería mi TRANSCRIPTOMA DE REFERENCIA, que es donde van a estar contenidos todos los READS y por ende todos los genes que yo alcance a secuenciar. Entonces este TRANSCRIPTOMA DE REFERENCIA se puede anotar y ¿Como se hace la anotación?. Se anota básicamente comparando estas secuencias, con las secuencias de base de datos del NCBI https://www.ncbi.nlm.nih.gov/pubmed/donde me puede decir que ésta secuencia o éste CONTIG se parece a una secuencia por ejemplo del PSI, o PSII. Tenemos otra base de datos que es más curada como el KEGG o el GO, asignándoles algún tipo de guión que es el GENEONTOLOGY.
El Blast2go es un programa de pago, sirve más que todo para anotar con una linea de comando dando una base de datos, lo bueno del blast2go nos hace un diagrama de flujo, el cual nos permite hacer una anotación funcional de genes o ensambles, separamos el transcriptoma de referencia, analizamos los diferentes genes que están expresados por categorías de los procesos biológicos, evaluando cuántos de estos genes corresponden a diferentes procesos.
Los READS que conforman el CONTIG, se normalizan y mapean. Mapear significa contar los READS, teniendo mi transcriptoma de referencia, quiero ver cuantos de los READS se alinean con algunos de los genes de una base de datos, para eso ocupo el programa Bowtie2 el que cuenta los READS, dándo una matriz de conteo.
¿Como se detecta Up-regulated o Down regulated?
El programa dice cuales son los Up o Down, tambien eso se puede ver con el Log2 Fold Change higher than 2 negativo generalmente dando una expresión positiva, por si hacemos la operación matemática.
Cuando usamos el Blast para las anotaciones, sólo para un gen demora mucho tiempo, imaginemos ¿Cuánto se demoraría para varios genes? Es por eso que es recomendable bajar toda la base de datos del Blast, entonces yo puedo comparar toda la base de datos completa en lo mismo que se demora usando la web del Blast.
En cuanto a costos un transcriptoma no baja de 3000000 de pesos chilenos. 400000000 de READS es lo que cuesta más o menos 2000000 de pesos chilenos. Estos 400000000 de READS los podemos ocupar de acuerdo a nuestras necesidades. Es decir, si tenemos el t0, t1, t2 y t3, lo más probable es que por cada uno de estos tiempos, se representa como 100000000 de READS.
En cada muestra se podría tener 35000000 READS aproximadamente, esto es lo que se conoce como "Profundidad de secuenciación" que significa la cantidad de READS que tengo disponible para la secuenciación en un tiempo y una condición.
Según los análisis estadísticos, es sobre 25000000 READS por muestra, siendo suficiente para capturar casi toda la muestra. Es decir, sobre 35000000 READS, nos aseguramos que tenemos sobre el 95% de los transcritos del organismo secuenciado. Concluyendo que el servicio que se pide es una "TRANSCRIPTÓMICA CON ANÁLISIS BIOINFORMÁTICO", esto entrega la expresión diferencial de los cluster, la cuantificación, la anotación, entonces nos va decir a que corresponden los CONTIG, entonces si se tiene los genes asociados, nos entregará la lista de todos los genes que tenemos en el Blast, KEGG o GO.
(FUENTE: Apuntes de sesiones teórico-práctico de pasantia doctoral del Student PhD. Ronald Huarachi Olivera en la Universidad de Chile, Pontificia Universidad Católica de Chile, Universidad de Santiago de Chile y Universidad Andrés Bello).
12.- BIOLOGÍA CIRCADIANA ¿COMO FUNCIONA EL RELOJ CIRCADIANO?:
Por ejemplo, si sellamos una habitación completamente y yo no sé lo que está ocurriendo afuera, igual uno se acostaría en el horario de afuera que se encuentra en oscuridad y despertará de manera autónoma más o menos a la hora que está saliendo el sol, que no lo veo, a eso se refiere el ritmo circadiano a algo ENDOGENO regulado por relojes moleculares. Si no lo hago así, me despierto cuando esta saliendo el sol, ahí estaría respondiendo a estímulos ambientales y no seria un RITMO CIRCADIANO, Por tanto, desde el punto de vista molecular, se esperaría a que si realizamos el silenciamiento de algunos de los componentes del reloj circadiano de la linea biológica, se pierda la sincronía del reloj celular. Una de las características del reloj circadiano es que puede funcionar en condiciones ambientales constantes. Si observamos que ocurre sólo cuando se tiene un fotociclo, entonces estaríamos tratando de un fenómeno manejado por señales ambientales.
Cuando nos referimos a que los ritmos circadianos son entrenables por la luz, nos indica que entrenar significa ordenar, si ustedes se van a Tokio mañana van a despertar a la hora de Arequipa al otro día, pero de poco andar mas o menos 1día/hora de fase. Es decir en dos semanas va a estar en la hora de Tokio, van a estar mas o menos sincronizados, eso quiere decir que el reloj puede cambiar de fase, su posición de inicio del día, según el horario del día donde estén, eso significa que son entrenables y pueden ser entrenables por la luz o por la temperatura.
Y la gracia que tienen los ritmos circadianos es coordinar los ritmos de sueño y vigilia, de fisiología y metabolismo con el ciclo de luz/oscuridad de la tierra. Los ritmos circadianos están en todas partes: hongos(crecimiento), plantas(ritmos de floración, movimiento de hojas), insectos, peces, aves, mamiferos(sueño y vigilia), incluso algunas bacterias.
FORMAS DE ESTUDIAR LOS RITMOS CIRCADIANOS: Hay muchos experimentos, que son más complejos que es jugar con días (alargar o disminuir), no con largos fotociclos, entonces ahí uno puede tener por ejemplo, lo siguiente: T24(8h de luz/16h de oscuridad), T16(8 h de luz/8 h de oscuridad) o T28(8h de luz/20h de oscuridad). Entonces a partir de esos tratamientos por asi decirlo, podemos evaluar lo siguiente: ¿Que va pasar con peak de G1? Va seguir ocurriendo siempre a la misma hora, se puede calcular la fase en la cual está ocurriendo con el peak de G1, comparando con el largo total del ciclo y en base a eso, uno puede inferir si hay un fenómeno que depende de la regulación interna del ritmo circadiano, o si simplemente es una respuesta preestablecida a los cambios ambientales, pero desde el punto de vista experimental es más complejo, se tendría que estar evaluando con citometría de flujo a distintas horas, idealmente no más de 4 horas. Se tendría que incubar la línea biológica en distintas condiciones, haciéndolo crecer lo que se denomina "entrenamiento de organismo" y luego por ejemplo al tercer día empezaría a evaluar por ejemplo cada 4 horas su comportamiento por citometría de flujo en los siguientes tratamientos(T24, T16 y T28).
